Recientemente se publicaba un estudio en Nature en el que se anunciaba la identificación de 83 variaciones del genoma relacionadas con la altura de las personas que las portaban. Es decir, los individuos con algunas de estas variaciones en su ADN tendían a ser más altos que los que no las poseían. Este estudio, llevado a cabo con unas 700 000 personas, también permitió conocer mejor cómo interaccionan los distintos mecanismos celulares relacionados con el crecimiento y, por tanto, ayudaría a comprender las enfermedades del desarrollo. Este tipo de análisis que buscan variantes genéticas que pueden estar relacionadas con alguna enfermedad son cada vez más comunes, pero ¿cómo se llevan a cabo?
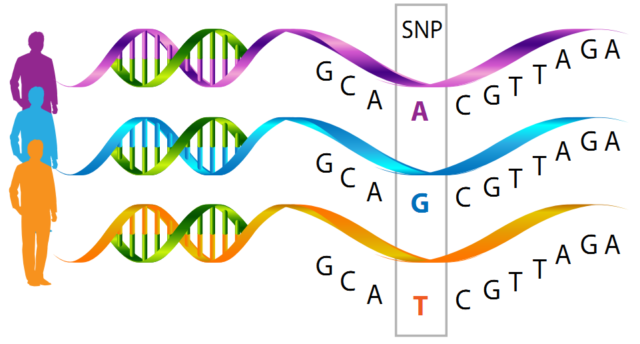
Los genomas de los seres humanos, aunque prácticamente idénticos entre sí, presentan pequeñas diferencias que son observables en los fenotipos (lo cual hace que podamos distinguirnos unos de otros, por ejemplo) y, en algunas ocasiones, pueden aumentar la predisposición a sufrir enfermedades complejas. Es bastante habitual que estas diferencias se presenten en una posición concreta de la cadena de ADN implicando a un único nucleótido, lo que se conoce como polimorfismo de un solo nucleótido (SNP, single nucleotide polymorphism). Recuerda bien estas siglas porque las utilizaremos con frecuencia a lo largo de este texto. En la siguiente imagen se representa un SNP con un nucleótido diferente en tres individuos.
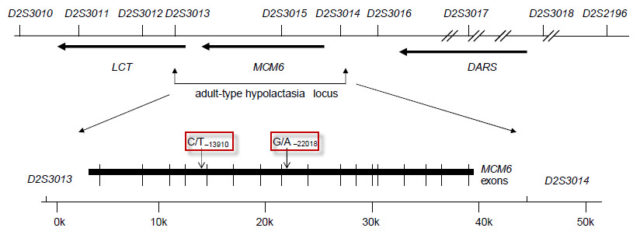
Por ejemplo, los SNPs situados a 13 910 y 22 018 bases del gen que codifica la lactasa son responsables de la intolerancia a la lactosa en adultos, en concreto las variantes con los nucleótidos citosina (C) y guanina (G) en ambos alelos, respectivamente.
Cabe destacar la diferencia entre SNP y mutación genética: para que una mutación concreta sea considerada SNP debe aparecer en al menos un 1% de la población. Se calcula que hay unos diez millones de SNPs en nuestro genoma. La mayor parte de estas variantes no se encuentra en las regiones codificantes de los genes (las que incluyen la información para, por ejemplo, fabricar proteínas), por lo que, en caso de afectar a algún rasgo fenotípico, lo harían probablemente a través de la expresión génica, es decir, al control de qué genes se activan en cada célula y en qué medida.
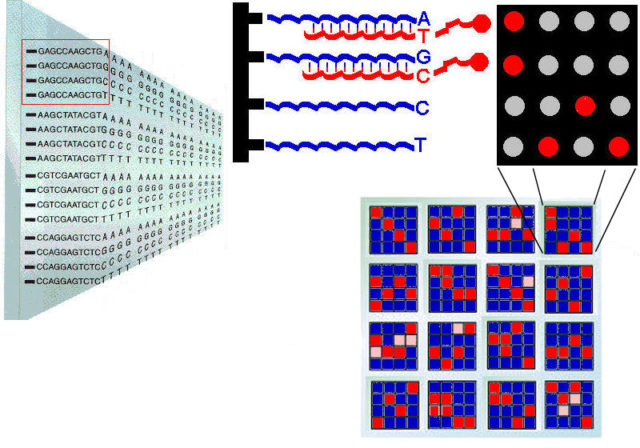
Con los avances en genotipado y secuenciación de ADN, el crecimiento de las bases de datos de genomas y las nuevas técnicas bioinformáticas, es posible realizar estudios a gran escala para analizar cómo influyen en determinadas enfermedades las diferencias concretas entre genomas de distintos individuos. Con chips de ADN de genotipado (SNP-chips) se puede averiguar rápidamente qué nucleótidos ocupan varios cientos de miles de posiciones concretas del genoma de una persona, justamente las posiciones asociadas a algunos de los SNPs conocidos.
En un estudio de asociación del genoma completo (GWAS, Genome-wide association study) se busca relacionar variaciones en el genoma (en concreto, uno o varios SNPs) con rasgos fenotípicos concretos. En particular, estos estudios buscan la predisposición genética a sufrir un determinado trastorno complejo. Deja que lo explique mejor: aunque una variante genética no sea directamente responsable de una enfermedad puede influir en nuestra predisposición a padecerla.
Para llevar a cabo estos estudios se selecciona a un grupo de personas con la enfermedad o trastorno a estudiar y un grupo (control) de individuos que no sufran esta patología. Se toma una muestra de ADN de todos los participantes del estudio y se analizan las variantes de cientos de miles de SNPs. En el caso de que se localice una variante concreta de uno o varios SNPs que sea mucho más abundante en el grupo de enfermos que en el grupo de control, se dice que es una variante asociada a la enfermedad. Veámoslo en un ejemplo sencillo.
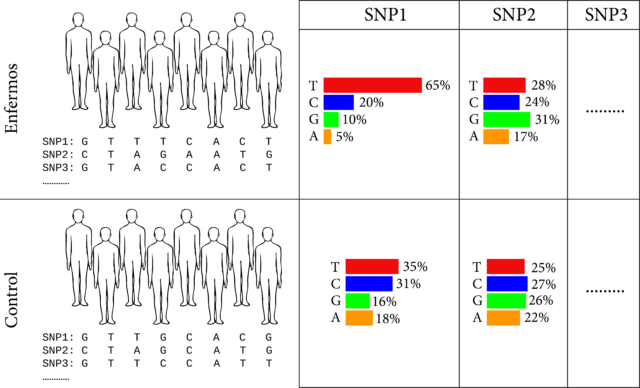
En la imagen anterior tendríamos un ejemplo de cómo se lleva a cabo un estudio de asociación genómica. Se toma un grupo de enfermos diagnosticados con una patología o trastorno y se obtienen sus SNPs. Se repite el procedimiento con un grupo de control (no diagnosticados) y se comparan las variantes de SNPs en el grupo control y de enfermos. En la figura se observa, por ejemplo (y a falta de hacer un análisis estadístico adecuado), que en el SNP1 hay una preponderancia de timinas en el grupo de enfermos respecto al grupo control, lo cual hace pensar que la presencia de una T en esta posición estaría asociada a la enfermedad bajo estudio.
Pero incluso aunque tal asociación exista, la realidad es más compleja. En el ejemplo anterior se observa que en el grupo de control hay un gran número de individuos que lleva una timina en el SNP1 pero no presenta la enfermedad. Y personas en el grupo de enfermos que no tienen timina en el SNP1. Esto ocurre porque puede que haya más SNPs implicados, mutaciones poco frecuentes no consideradas o que influyan factores ambientales.
En resumen, si se descubre que una variante genética concreta de la población está correlacionada con un rasgo o patología se dice que ambos están asociados (aun cuando esta asociación no implique causalidad). Por ejemplo, con uno de los primeros GWAS se descubrió que los individuos que poseían una de entre cinco variaciones concretas de tipo SNP aumentaban entre dos y tres veces la predisposición a padecer degeneración macular asociada a la edad.
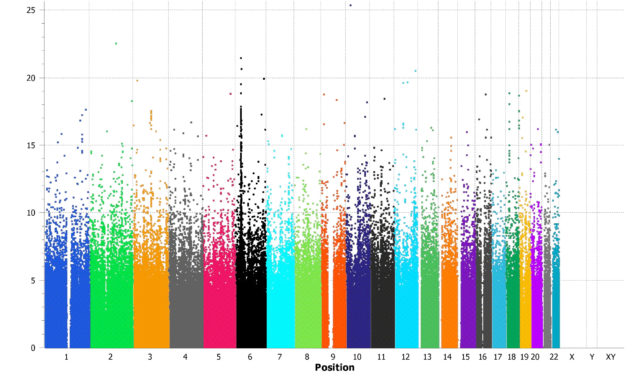
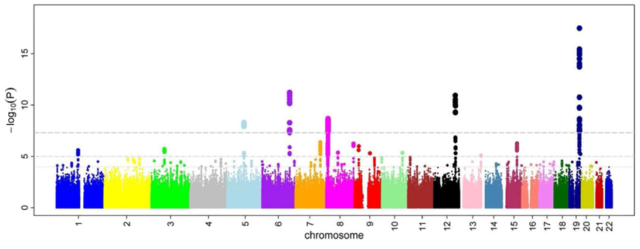
Los resultados de los GWAS se suelen mostrar con un diagrama de Manhattan, en el que se representa, agrupada por posiciones en cromosomas, la relevancia estadística de cada SNP: valores más altos indican una mayor asociación de la posición en el genoma con el riesgo de padecer la enfermedad bajo estudio. Por ejemplo, este es el diagrama de Manhattan de un estudio en el que se buscaban SNPs asociados a problemas de microcirculación descritos en base al calibre vascular de los vasos de la retina. En él se observa como hay cinco SNPs (los que superan un determinado umbral estadístico indicado con una línea punteada) que podrían estar asociados a esta patología.
Que encontremos SNPs asociados a enfermedades no implica que sepamos cuáles son los mecanismos que desencadenan el problema. Para ello hay que analizar con más detalle la zona del genoma donde se encuentra el SNP y así detectar a qué gen puede afectar, o tratar de relacionarlo con cambios epigenéticos o de expresión génica, y esto no siempre es fácil. Pero de algunos de los GWAS realizados sí que se han obtenido resultados útiles para tratar algunas enfermedades. Por ejemplo, en el caso antes citado de la degeneración macular asociada a la edad se encontraron algunos SNPs asociados a la enfermedad que sugirieron que esta podría estar relacionada con un proceso inflamatorio, por lo que actualmente se están explorando tratamientos antiinflamatorios para esta patología.
Desde este primer estudio se han encontrado asociaciones de SNPs con varias patologías como la diabetes de tipo 2, enfermedades coronarias, Crohn y varios tipos de cáncer como los de esófago, pulmón y páncreas, entre otras. En la fecha de publicación de este artículo se habían realizado cerca de 3 000 GWAS y encontrado asociaciones de enfermedades con más de 30 000 SNPs. En el siguiente diagrama (que puedes encontrar completo y ampliable aquí) se representan las asociaciones más relevantes de enfermedades con SNPs de algunos cromosomas.
Una de las aplicaciones de los GWAS a patologías y trastornos concretos es la evolución a una medicina personalizada en la que se tengan en cuenta los mecanismos específicos que provocan la enfermedad para proporcionar un tratamiento adecuado a las características particulares de un paciente. Actualmente ya existen compañías que permiten obtener las variantes SNP concretas de un individuo. Por ejemplo, la empresa 23andme ofrece el análisis de unos 600.000 SNPs mediante una muestra de saliva desde tan solo 99 dólares, aunque de todos estas variantes únicamente unas 25.000 están actualmente asociadas a rasgos específicos. En España no está accesible este servicio, aunque la empresa valenciana tellmeGen ofrece un producto similar (pese a que no informa del número de SNPs analizados).
Sin embargo, y a pesar del interés de los estudios de asociación de variaciones genéticas a rasgos observables, existen muchas dudas de su utilidad a la hora de predecir futuras enfermedades. Salvo algunas excepciones notables, los SNPs asociados a rasgos concretos presentan efectos muy pequeños en la predisposición a padecer una enfermedad, por lo que su utilidad en el pronóstico es limitada. Por ejemplo, menos de la cuarta parte de la variabilidad genética de la enfermedad de Crohn se debe a los SNPs asociados más relevantes, por lo que debe haber otros efectos que se nos escapan, como variantes genéticas tan poco frecuentes que no se consideran como SNPs.
Además, aunque se han identificado muchas variaciones genómicas asociadas a enfermedades, los mecanismos de acción subyacentes de la inmensa mayoría siguen siendo desconocidos y esto limita la utilidad de estos análisis de asociación. Sólo un 10% de los SNPs asociados a rasgos se encuentran en secuencias de ADN que codifican proteínas. Casi la mitad de las asociaciones encontradas se encuentran fuera de los genes, por lo que su papel en la enfermedad se debería a su relación con la expresión génica. Una vía para comprender cuál es la influencia de los SNPs en los rasgos asociados pasa por analizar cómo afectan a la expresión génica en tejidos concretos mediante estudios de interrelación con el transcriptoma y epigenoma. Este tipo de estudios ya han empezado a realizarse y nos podrían ofrecer una visión más exacta de por qué estamos más o menos predispuestos a padecer algunas enfermedades.
Este post ha sido realizado por Guillermo Peris (@Waltzing_piglet) y es una colaboración de Naukas con la Cátedra de Cultura Científica de la UPV/EHU.
Referencias
- Genomewide Association Studies and Assessment of the Risk of Disease. Manolio, T. A. (2010). N Engl J Med 363:166-176. doi: 10.1056/NEJMra0905980
- Finding Genes for Common Diseases Using GWAS. Bowcock, A. M. (2015). Nature Education 8(5):5.
- Rare and low-frequency coding variants alter human adult height. Marouli, E. et al (2017). Nature 542(7640):186-190. doi: 10.1038/nature21039.
Enlaces 16//6//2017
[…] operativo escrito en ADN (ArsTechnica) // Sobre la predosposición genética a tener enfermedades (CdCC) // Sobre la Palinología, la ciencia que estudia el polen y las esporas (Wiki 1 y 2) // Sobre […]
Sobre la predisposición genética a padecer enfermedades (II) – Cuaderno de Cultura Científica
[…] un artículo anterior hablábamos de cómo se descubren las variantes genéticas (los polimorfismos de un solo […]
Descifrando rostros a partir de genes – El Loco de la Colilla
[…] asociación del genoma completo (GWAS, Genome-wide association study, lo expliqué con más detalle aquí) y nos pueden señalar qué variantes concretas están asociadas con el rasgo que tratamos de […]
Surströmming, jamón de Jabugo y CRISPR – El Loco de la Colilla
[…] un estudio de asociación genómica realizado en Islandia y publicado en la revista Current Biology (y que podéis encontrar más […]
Los límites de los test de ADN para la predicción de enfermedades — Cuaderno de Cultura Científica
[…] Sobre la predisposición genética a padecer enfermedadesLas bases genéticas de las enfermedades digestivas […]