Un método para la determinación rápida de especies bacterianas por su código de barras
 Este texto es una colaboración del Cuaderno de Cultura Científica con Next
Este texto es una colaboración del Cuaderno de Cultura Científica con Next
Una parte de la resistencia a los antibióticos de las bacterias patógenas se debe al uso de antibióticos de amplio espectro en las primeras etapas de la infección porque no se ha conseguido determinar qué especie concreta es la causante. Ahora una nueva herramienta desarrollada por el equipo de investigadores que encabeza Lucienne Otten, de la Universidad de Warwick (Reino Unido), promete acelerar la determinación de la especie correcta. El método es barato y accesible en el punto de tratamiento (la consulta del médico o la casa del enfermo).
La resistencia a los antibióticos es una de las problemas de salud más importantes a los que se enfrenta la Humanidad en las próximas décadas. Y se está haciendo cada vez mayor debido a la ausencia de nuevos fármacos y de herramientas de diagnóstico en los puntos de atención al paciente que aseguren que se usa en cada caso el mejor fármaco disponible.
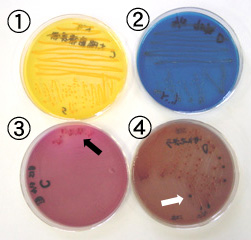
Los métodos de diagnóstico actuales para identificar a las bacterias requieren, bien cultivarlas para obtener una densidad mayor, algo que es muy lento, o bien tecnologías de secuenciación de ADN que son más rápidas, pero que requieren horas, un equipamiento específico no portátil y personal especializado en usarlo.
El método de Otten et al. es muy ingenioso. Se basa en que muchas bacterias se adhieren a las células mediante unas estructuras químicas de su superficie llamadas adhesinas. Como cada tipo de bacteria tiene un patrón de adhesinas característico muy concreto, cada una se une con una fuerza diferente a cada una de las superficies celulares que tienen unos azúcares (glicanos) también característicos.
Si ahora se toma una placa plástica con distintos huecos, y en cada uno de esos huecos se inmovilizan glicanos que simulen distintos tipos celulares, cada bacteria se unirá a los glicanos de cada hueco con una fuerza diferente. Comparando los datos de las distintas especies bacterianas, resulta una especie de código de barras para cada una.
Una vez que se ha construido una biblioteca de códigos de barras es posible identificar cualquier muestra desconocida de bacteria con solo depositarla en la placa plástica y ver qué huecos “dan positivo”, proporcionando de esta manera su código de barras; siempre y cuando esté en la base de datos, obviamente.
Este método analítico no solo es más rápido que los existentes sino también mucho más barato. Los investigadores afirman en el artículo que publican en Molecular BioSystems, y que es de acceso gratuito, que ya están trabajando en un kit de diagnóstico desechable para la determinación rápida de especies bacterianas, el diagnóstico de resistencias bacterianas e incluso la monitorización de brotes epidémicos.
Referencia:
L. Otten et al (2016) Discrimination between bacterial species by ratiometric analysis of their carbohydrate binding profile Mol. BioSyst. DOI: 10.1039/C5MB00720H
Sobre el autor: César Tomé López es divulgador científico y editor de Mapping Ignorance


Peñaloza Verónica
Gracias!
BigBenCentre
Que pasada, ojalá y estos avances estén pronto al alcance de todos.
Un método para la determinación r…
[…] Una parte de la resistencia a los antibióticos de las bacterias patógenas se debe al uso de antibióticos de amplio espectro en las primeras etapas de la infección porque no se ha conseguido determinar qué especie concreta es la causante. […]
Un método para la determinación rápida de especies bacterianas por su código de barras
[…] Un método para la determinación rápida de especies bacterianas por su código de barras […]
Hitos en la red #98 20D Edition – Naukas
[…] cohete con el que Elon Musk quiere conquistar Marte por Daniel Marín. Y por eso son interesantes Un método para la determinación rápida de especies bacterianas por su código de barras o Del cerdo, hasta la hidroxiapatita. Y por eso se han comercializado con […]