Del barro al ADN: cómo el ADN ambiental puede revelar la función de los microbios en los estuarios

Los estuarios y marismas costeras son verdaderos nodos de biodiversidad. En estos ecosistemas dinámicos convergen ambientes terrestres, marinos y de agua dulce. Además de su riqueza ecológica, prestan servicios cruciales: sirven de criaderos para peces, protegen frente a tormentas, depuran contaminantes y aportan valor recreativo [1].
Sin embargo, muchos estuarios están en peligro. La contaminación urbana, agrícola e industrial, el cambio climático y, sobre todo, la pérdida directa de hábitat por la urbanización y el relleno de tierras están dejando huella [1-3]. Para proteger estos ecosistemas es esencial saber cómo están cambiando, y eso requiere biomonitoreo: seguir su estado de salud a lo largo del tiempo.
Microbios: los invisibles que sostienen los ecosistemas estuarios
Tradicionalmente, el monitoreo se ha basado en la identificación visual de organismos del sedimento (gusanos, almejas, crustáceos), un proceso costoso y que requiere especialistas taxonómicos. Pero los avances en biología molecular están ofreciendo alternativas prometedoras, como el análisis de ADN ambiental (eDNA): fragmentos de material genético que los organismos dejan en el entorno y que permiten conocer qué seres vivos han pasado por allí [4-6].
El eDNA ha revolucionado especialmente el estudio de los microorganismos. Aunque invisibles, tienen un papel central en los ciclos biogeoquímicos globales y en procesos locales como la depuración de contaminantes o el reciclaje de nutrientes [7,8]. También están detrás de muchas consecuencias negativas cuando un ecosistema se deteriora: proliferación de algas tóxicas, emisiones de metano, etc.
No solo quién está, sino qué hacen
Aun así, los microbios rara vez se incluyen en los programas de biomonitoreo, y cuando lo hacen, se tratan como cualquier otro organismo: identificándolos mediante bases de datos de secuencias de referencia y utilizando conocimientos previos sobre qué organismos son sensibles o están asociados con diferentes tipos de perturbación. Así, se infiere la salud ambiental a partir de la presencia o ausencia de especies indicadoras [5,9].
Pero esto apenas rasca la superficie. El eDNA no solo nos dice «quién está ahí», también permite estudiar qué funciones metabólicas realizan esos microbios. Los genes que codifican enzimas implicadas en la digestión de materia orgánica, la producción de gases o la transformación de compuestos químicos nos hablan de cómo funciona el ecosistema, no solo de su composición.
Además, los microbios interactúan entre sí: pueden depender unos de otros (simbiosis) o competir por recursos. Si sabemos qué genes funcionales están presentes, podemos inferir procesos ecológicos más complejos [10].
Tres caminos para estudiar la función microbiana
En un estudio reciente en estuarios de la costa vasca, Abad-Recio y colaboradores [6] compararon tres enfoques distintos para analizar funciones microbianas a partir del eDNA:
- Metagenómica
Es el método más completo: se secuencia directamente todo el ADN de la muestra. Permite identificar tanto las especies presentes como sus genes funcionales. Técnicamente es accesible, pero los datos son tan masivos que analizarlos es todo un reto, dada nuestra limitada comprensión de la diversidad microbiana [11]. - Inferencia funcional a partir de metabarcoding
Aquí se amplifica un gen marcador (normalmente 16S rRNA) para saber qué microbios hay, y luego se usa el conocimiento previo sobre sus funciones típicas. En el estudio, se utilizó la herramienta bioinformática Tax4Fun [12]. Es un enfoque más barato y rápido, aunque sus resultados funcionales son indirectos. - Microarrays funcionales (GeoChip)
En lugar de secuenciar, se usa una «placa» con sondas de ADN que detectan genes específicos mediante hibridación fluorescente [13]. Es económico y los datos se analizan más fácilmente. Sin embargo, está limitado por el número y tipo de genes que se pueden incluir en el chip, lo que impide capturar toda la diversidad funcional del entorno.

¿Cuál funciona mejor?
El equipo analizó muestras de sedimento de ocho sitios: cuatro contaminados (metales pesados, PCBs o exceso de nutrientes) y cuatro relativamente intactos.
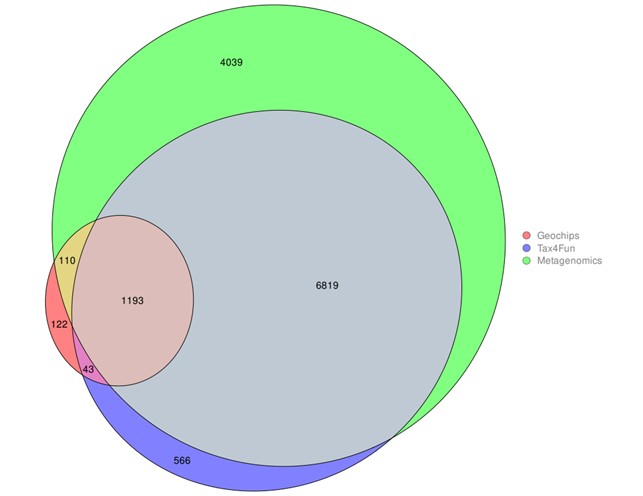
- La metagenómica detectó la mayor diversidad de genes, seguida del metabarcoding. GeoChip detectó muchos menos genes, como era previsible por sus limitaciones técnicas (Figura 2) [6].
- Aunque hubo solapamiento entre métodos, las abundancias relativas de los genes fueron más consistentes entre metagenómica y metabarcoding, mientras que GeoChip dio resultados más discordantes.
- En cuanto a genes funcionales asociados a la contaminación, el metabarcoding identificó más de 2300 genes, frente a 655 con metagenómica y solo uno con GeoChip. Los genes identificados estaban relacionados con procesos típicos de ambientes degradados: metanogénesis, fermentación anaeróbica y reducción disimilatoria de sulfato.
¿Qué nos dice todo esto?
La metagenómica y la inferencia funcional a partir de metabarcoding ofrecen herramientas muy potentes para integrar a los microbios en el monitoreo ambiental. Pueden ayudar a detectar signos tempranos de degradación o predecir emisiones perjudiciales como metano o ácido sulfhídrico. En cambio, el método GeoChip parece más limitado, aunque útil en contextos específicos.
Pese a su enorme potencial, ningún programa de monitoreo ambiental rutinario ha incorporado aún estas técnicas. Uno de los principales obstáculos es la complejidad del análisis bioinformático necesario para extraer información útil de los datos [6].
Pero lo que está claro es que saber qué microbios están presentes ya no es suficiente. Conocer también qué funciones realizan puede marcar la diferencia a la hora de proteger los ecosistemas de los que dependemos.
Referencias
[1] Boerema, A. & Meire, P. (2017). Management for estuarine ecosystem services: A review. Ecological Engineering, 98, 172–182.
[2] Lanzén, A. et al. (2021). A microbial mandala for environmental monitoring – predicting multiple impacts on estuarine prokaryote communities of the Bay of Biscay. Molecular Ecology, 30, 2969–2987.
[3] DiBattista, J. D. et al. (2024). Tree of life metabarcoding can serve as a biotic benchmark for shifting baselines in urbanized estuaries. Environmental Research, 258, 119454.
[4] Cordier, T. et al. (2018). Embracing Environmental Genomics and Machine Learning for Routine Biomonitoring. Trends in Microbiology, 27, 387–397.
[5] Cordier, T. et al. (2020). Ecosystems monitoring powered by environmental genomics: a review of current strategies with an implementation roadmap. Molecular Ecology, 30, 2937–2958.
[6] Abad-Recio, I. L. et al. (2024). Toward functional profiling for eDNA-based monitoring in coastal environments: A comparison of three approaches. Environmental DNA, 6, e504. doi: 10.1002/edn3.504
[7] Hugerth, L. W. & Andersson, A. F. (2017). Analysing Microbial Community Composition through Amplicon Sequencing. Front Microbiol, 8, 1561.
[8] Falkowski, P. G. et al. (2008). The microbial engines that drive Earth’s biogeochemical cycles. Science, 320, 1034–1039.
[9] Aylagas, E. et al. (2017). A bacterial community-based index to assess the ecological status of estuarine and coastal environments. Marine Pollution Bulletin, 114, 679–688.
[10] Codello, A. et al. (2022). Microbial co-occurrence networks as a biomonitoring tool for aquatic environments: a review. Mar. Freshwater Res.
[11] Liu, S. et al. (2025). Analysis of metagenomic data. Nature Reviews Methods Primers, 5.
[12] Wemheuer, F. et al. (2020). Tax4Fun2: prediction of habitat-specific functional profiles and functional redundancy based on 16S rRNA gene sequences. Environmental Microbiome, 15.
[13] He, Z. et al. (2007). GeoChip: a comprehensive microarray for investigating biogeochemical, ecological and environmental processes. The ISME Journal, 1, 67–77.
Sobre el autor: Anders Lanzén, Investigador Senior en AZTI (Marine Research, BRTA), Pasaia, Gipuzkoa, y Research Associate Professor en IKERBASQUE – Basque Foundation for Science, Bilbao, Bizkaia.
Sobre AZTI: El propósito de AZTI es impulsar un cambio positivo para el futuro de las personas, contribuyendo a una sociedad saludable, sostenible e íntegra. Especializado en el medio marino y la alimentación, AZTI aporta productos y tecnologías de vanguardia y de valor añadido basados en ciencia e investigación sólidas. AZTI es miembro de Basque Research and Technology Alliance (BRTA).
Basque Research & Technology Alliance (BRTA) es una alianza que se anticipa a los retos socioeconómicos futuros globales y de Euskadi y que responde a los mismos mediante la investigación y el desarrollo tecnológico, proyectándose internacionalmente. Los centros de BRTA colaboran en la generación de conocimiento y su transferencia a la sociedad e industria vascas para que sean más innovadoras y competitivas. BRTA es una alianza de 17 centros tecnológicos y centros de investigación cooperativa y cuenta con el apoyo del Gobierno Vasco, SPRI y las Diputaciones Forales de Araba, Bizkaia y Gipuzkoa.

