Posible estrategia terapéutica contra la esclerosis lateral amiotrófica
La esclerosis lateral amiotrófica (ELA) es una enfermedad neurodegenerativa que afecta a las neuronas del cerebro y la médula espinal provocando la pérdida del control muscular. Un estudio liderado por la Universidad de Barcelona y el Centro de Física de Materiales (CFM, mixto CSIC-UPV/EHU) ha diseñado una potencial estrategia terapéutica para abordar esta patología que todavía no tiene cura. Se trata de una trampa molecular, que evita que uno de los compuestos peptídicos causantes de la ELA genética más común -el dipéptido polyGR- provoque sus efectos tóxicos en el organismo. Los resultados muestran que esta estrategia reduce la muerte de las neuronas de pacientes y en un modelo animal (moscas del vinagre) de la enfermedad.
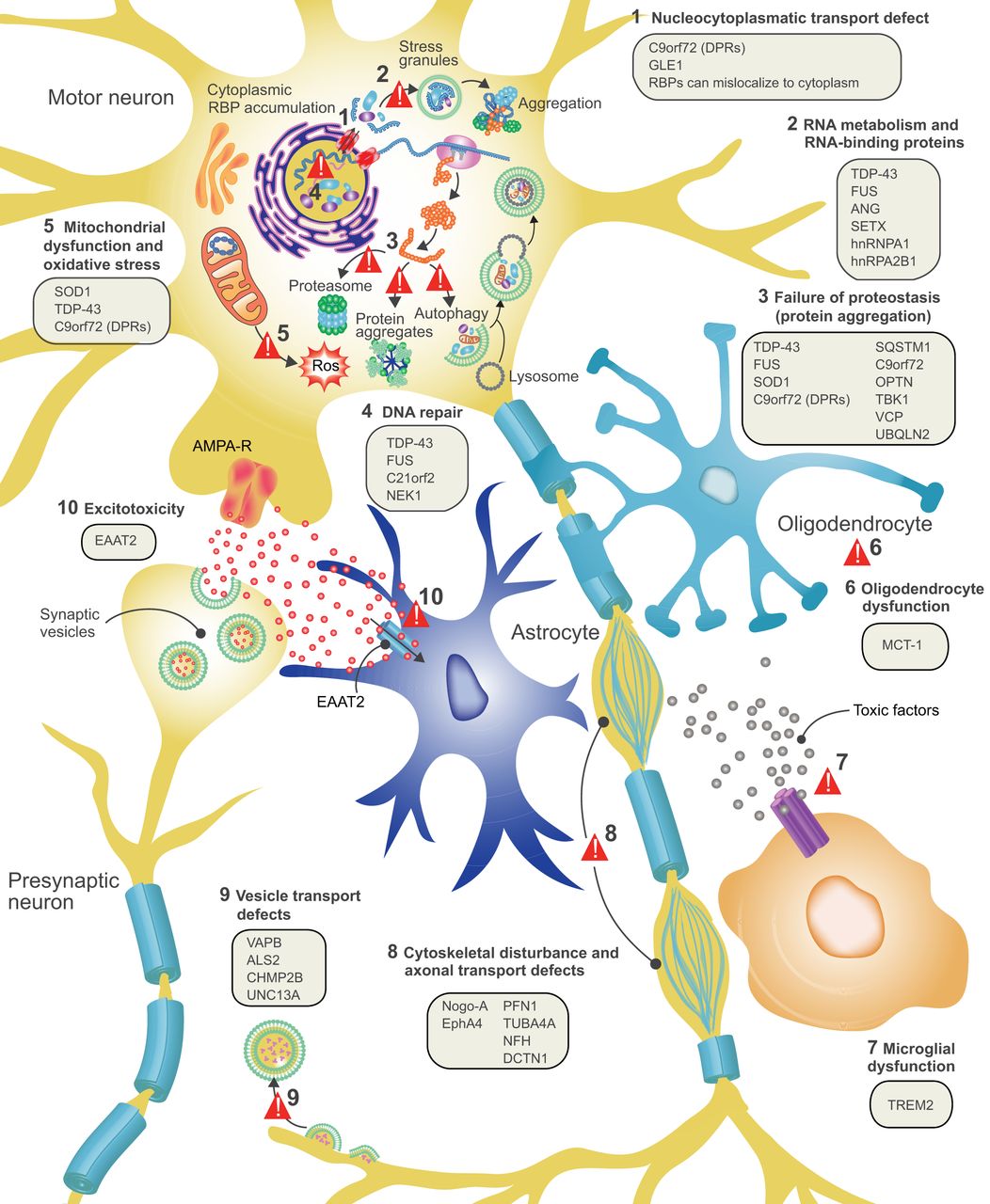
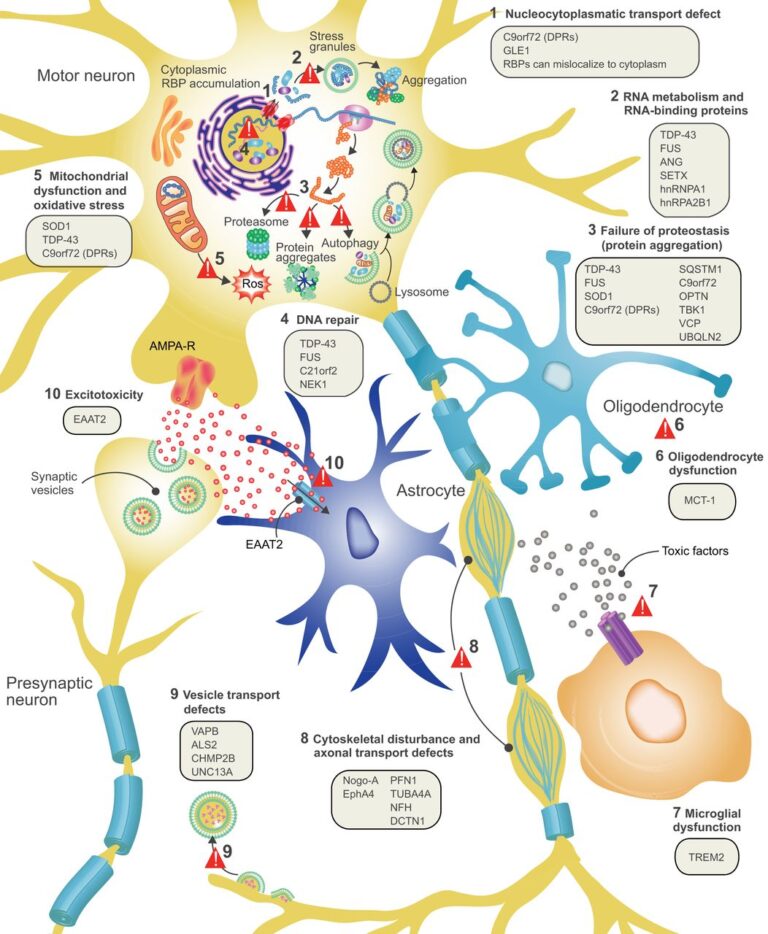
Una de las causas genéticas más frecuentes de la ELA es la mutación en el gen C9orf72, ya que se encuentra en aproximadamente el 33% de las personas pacientes afectadas por la ELA familiar y el 5% de las afectadas por la ELA esporádica en España. En estos/as pacientes se generan unos dipéptidos con gran cantidad de cargas positivas que producen efectos altamente tóxicos en las neuronas motoras. En la primera parte del estudio, el equipo de investigación combinó técnicas computacionales y experimentales para mejorar la comprensión molecular de estos dipéptidos y cómo producen este proceso patológico.
Una unión tóxica para las neuronas
Los resultados mostraron que la toxicidad de estos compuestos se debe en parte a lo que se unen al ARN ribosomal (ARNr), una molécula que participa en el proceso de traducción de la información genética y la síntesis de proteínas en la célula. «Hemos visto que estos dipéptidos, especialmente los ricos en el aminoácido arginina (poli-glicina-arginina o polyGR), se unen a una región concreta del ARNr afectando a la biosíntesis de ribosomas (pequeñas estructuras que se encargan de sintetizar las proteínas de nuestro organismo) y la traducción de proteínas en neuronas motoras humanas, produciendo la muerte de éstas», explica el profesor Juan Alberto Ortega Cano. «Además – añade el investigador – esta interacción de los polyGR con el ARNr es mucho más fuerte que la interacción del polyGR con otras proteínas ribosomales que se habían descrito previamente en otros estudios, y explica por qué estos dipéptidos tienen gran afinidad en unirse a los ribosomas de las células».
Ante estos resultados, el equipo de investigación diseñó una innovadora estrategia para engañar a los dipéptidos polyGR y reducir su toxicidad. Crearon una trampa, una molécula que imitaba la secuencia específica del ARNr con la que se unen los polyGr durante el proceso patológico, con el objetivo de evitar así los efectos neurotóxicos de esta unión. La aplicación de esta estrategia en neuronas derivadas de tejido de pacientes in vitro y en modelos de la enfermedad (moscas del vinagre) in vivo muestran que «reduce los defectos en la biosíntesis de ribosomas en la traducción de proteínas y la toxicidad en cielo que expresan polyGR, así como la muerte en motoneuronas de pacientes de ELA con mutaciones en el gen C9orf72», detalla el investigador.
Aunque todavía queda mucha investigación por validar y comprender completamente el funcionamiento de esta innovadora estrategia, los/las autores/as señalan en el artículo que estos prometedores resultados refuerzan la idea de que el uso de trampas de ARN es útil «no sólo para estudiar las interacciones ARN-proteína, sino también para proteger a las neuronas de los efectos perjudiciales de proteínas anómalas que se generan en otras enfermedades neurodegenerativas».
Referencia:
Ortega, J.; Sasselli, I.; Boccitto, M.; Fleming, A.; Fortuna, T.; Li, Y.; Sato, K.; Clemons, T.; Daley, E.; Nguyễn, T.; Anderson, E.; Ichida, J.; Pandey, U.; Wolin, S.; Stupp, S.; Kiskinis, E. (2023) CLIP-Seq analysis enables the design of protective ribosomal RNA bait oligonucleotides against C9ORF72 ALS/FTD poly-GR pathophysiology Science Advances doi: 10.1126/sciadv.adf7997
Edición realizada por César Tomé López a partir de materiales suministrados por UPV/EHU Komunikazioa