Cómo se forman los condensados dentro de las células
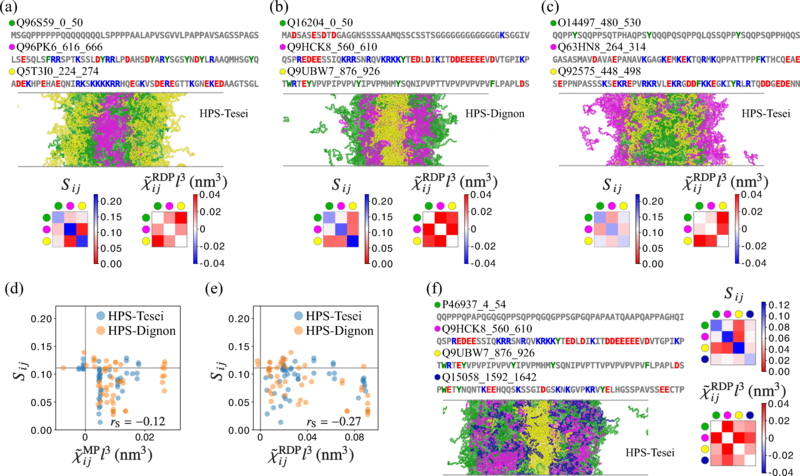
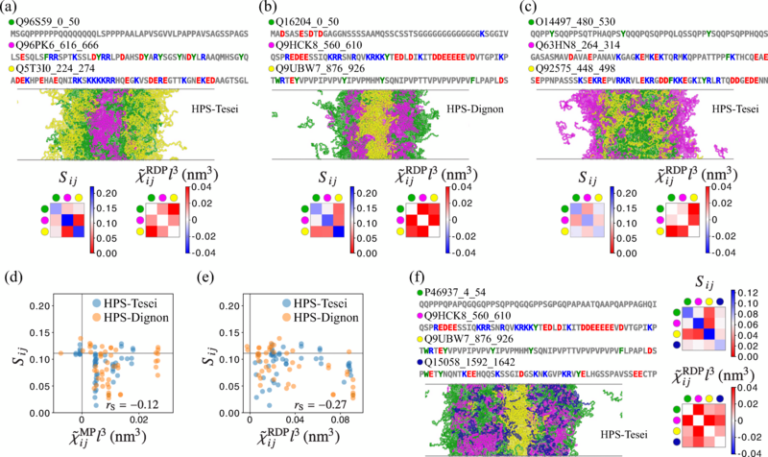
Las células biológicas contienen pequeñas gotas llamadas condensados que consisten en conjuntos de proteínas y otras moléculas. Estas gotas desempeñan papeles importantes en varias funciones celulares, como las reacciones bioquímicas. Se sabe que las interacciones entre las regiones intrínsecamente desordenadas (IDR, por sus siglas en inglés) de diferentes proteínas (partes de una proteína que carecen de una estructura bien definida) impulsan la formación de condensados. Pero los detalles exactos de cómo intervienen las IDR en esta formación no estaban claros.
Ahora Kyosuke Adachi y Kyogo Kawaguchi del Centro RIKEN para la Investigación de la Dinámica de Biosistemas en Japón han ideado un modelo que proporciona esa información. Los investigadores afirman que su modelo podría usarse para obtener otros conocimientos clave sobre los principios fundamentales de la organización celular.
Adachi y Kawaguchi han realizado simulaciones de la dinámica molecular de la formación de condensados para más de 200 IDR que se conocen en proteínas humanas. A continuación han combinado los resultados de estas simulaciones con una nueva aproximación para la dinámica de los IDR. Finalmente, han desarrollado una teoría que les permite vincular la fuerza de las interacciones entre los IDR de diferentes proteínas con el orden específico de los aminoácidos de los IDR, los componentes básicos de las proteínas.
El nuevo modelo indica que las IDR formadas por ciertas cadenas de aminoácidos tienen más probabilidades de unirse, lo que hace que sus proteínas se agrupen en un condensado. El modelo también muestra que las interacciones entre las IDR afectan a la posibilidad de que varios condensados puedan coexistir en una célula sin que se fusionen, y puede utilizarse para determinar cuántos condensados oexistentes puede albergar una célula.
Según los investigadores, el nuevo modelo podría ayudar a mejorar la comprensión de muchos procesos biológicos, incluidos los mecanismos por los que las células compartimentan sus funciones y la dinámica de las IDR de una proteína en diferentes condiciones.
Referencias:
Kyosuke Adachi & Kyogo Kawaguchi (2024) Predicting Heteropolymer Interactions: Demixing and Hypermixing of Disordered Protein Sequences Phys. Rev. X doi: 10.1103/PhysRevX.14.031011
Sobre el autor: César Tomé López es divulgador científico y editor de Mapping Ignorance