Detectar y destruir: bacterias equipadas con radar químico
No es la primera vez que nos ocupamos en esta sección de las extraordinarias habilidades desarrolladas por las bacterias. Hemos visto como son capaces de inventarse un nuevo gen para defenderse de los virus, o como se preparan para el invierno midiendo el fotoperiodo. Hoy nos ocuparemos de una bacteria que ha desarrollado un auténtico “radar químico” para detectar la presencia de amebas y elaborar una estrategia defensiva contra ellas. Las amebas, un grupo de protistas unicelulares, son uno de los principales enemigos de las bacterias, junto a virus y nematodos.

La bacteria de la que hablamos es Pseudomonas syringae, un bacilo flagelado, tristemente célebre por sus variantes patógenas para muchos cultivos vegetales y por las graves pérdidas económicas que causa (Figura 1). Un enemigo natural de estas bacterias es la ameba social Polysphondylium pallidum. Desde hace algún tiempo se conocía la existencia de cepas de P. syringae que resisten el ataque de las amebas. Poco a poco vamos conociendo los mecanismos de esta resistencia.
El grupo de Pierre Stallforth en la Universidad de Jena describió en 2023 unos lipopéptidos (cadenas cortas de aminoácidos unidas a lípidos) producidos por Pseudomonas y que eran toxicos para hongos y amebas. Llamaron a estas moléculas keanumicinas, por Keanu Reeves, aludiendo a la letalidad de su implacable personaje cinematográfico en la saga John Wick.
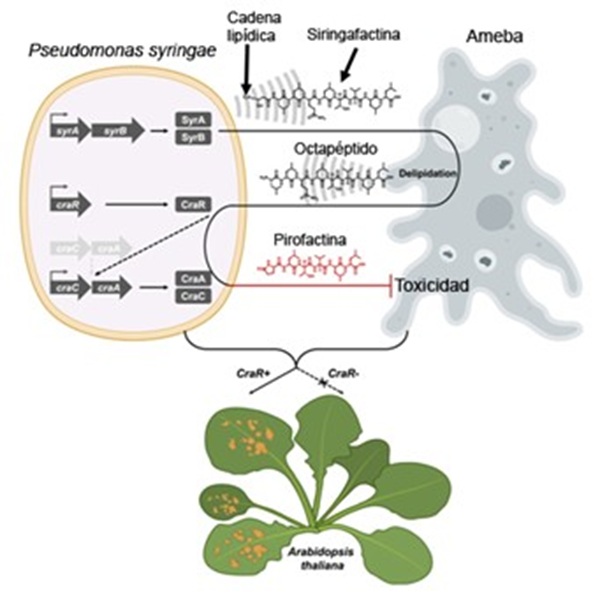
Este mismo equipo de investigación acaba de publicar en la revista Cell un artículo describiendo el sorprendente mecanismo que utilizan algunas cepas de P. syringae para detectar la proximidad de la ameba Polysphondylium. Se trata de un auténtico “radar químico”, como veremos a continuación (Figura 2).
El grupo de Stallforth comenzó por seleccionar dos cepas de P. syringae, una de ellas sensible a la depredación por las amebas (SZ57) y otra resistente a sus ataques (SZ47). Ambas tienen una gran similitud genética y son igualmente patógenas para las plantas. Las dos secretan lipopéptidos, las siringafactinas A y C, necesarias para que las bacterias desplieguen el comportamiento de agrupación y desplazamiento coordinado conocido como swarming en inglés. Las siringafactinas no son tóxicas para las amebas pero las absorben y las procesan, eliminando la porción lipídica y produciendo un octapéptido (cadena de ocho aminoácidos).
Los investigadores detectaron que la cepa resistente SZ47, pero no la SZ57, era capaz de procesar a su vez el octapéptido procedente de la ameba transformándolo en pirofactina, una molécula muy tóxica para las amebas. Esta era la clave de la protección de la cepa SZ47 contra la depredación. La conversión requería de un gen, llamado CraR (regulador del radar químico) presente en el genoma de SZ47, pero no en el de SZ57. La mutación de CraR en la cepa SZ47 la volvía vulnerable a la depredación por parte de las amebas.
La proteína expresada por el gen CraR se une al octapéptido y esta combinación induce la expresión de dos enzimas, CraA y CraC, responsables de la transformación del octapéptido en pirofactina.
Este mecanismo de defensa quedó perfectamente probado cuando se introdujo el gen CraR en la cepa CZ57, que a partir de ese momento se volvió inmune al ataque de las amebas. Tanto la cepa SZ47 como la CZ57 complementada con el gen CraR fueron capaces de infectar plantas sin importarles la presencia de amebas.
Lo sorprendente del sistema es su simplicidad y su economía. Un mismo tipo de molécula es utilizado para el desplazamiento de la bacteria (siringafactinas), para la detección de las amebas (octapéptido derivado de la delipidación de las siringafactinas) y para su destrucción (pirofactina derivada del octapéptido).
Estos resultados pueden ser útiles para controlar las infecciones producidas por P. syringae ya que la inhibición de la actividad de CraR dejaría a este fitopatógeno inerme frente a las amebas.
Referencias
Zhang, S., Schlabach, K., Pérez Carrillo, V.H. et al. (2025). A chemical radar allows bacteria to detect and kill predators. Cell. doi: 10.1016/j.cell.2025.02.033.
Sobre el autor: Ramón Muñoz-Chápuli Oriol es Catedrático de Biología Animal (jubilado) de la Universidad de Málaga.
