
Un trabajo de investigación llevado a cabo por el Grupo de Espectroscopía del Departamento de Química Física de la UPV/EHU y del Instituto Biofisika ha conseguido determinar con resolución atómica la estructura de los azúcares que forman parte del ADN, la 2-desoxirribosa. Se trata de “una resolución sin precedentes; hemos podido posicionar en el espacio cada uno de los átomos de este azúcar”, tal como describe el líder del grupo, Emilio J. Cocinero. El trabajo es portada de ACS Central Science.
Cocinero considera este resultado como la culminación de un trabajo que les ha llevado más de diez años: “Este resultado ha sido posible gracias al aumento de la sensibilidad del espectrómetro de microondas que tenemos en nuestro grupo, que hemos diseñado, construido y modificado nosotros mismos, y que ahora mismo está entre los 3 mejores aparatos de este tipo en el mundo”.
Una de las principales dificultades que tuvieron que superar fue la gran variabilidad y flexibilidad entre las diferentes formas o conformaciones que pueden adoptar las moléculas de 2-desoxirribosa. Los átomos que conforman estas moléculas de azúcar se pueden organizar formando anillos de cinco miembros, o formando anillos de seis miembros. “En la naturaleza, las formas biológicas presentan anillos de cinco miembros, pero en los experimentos, al aislar completamente el azúcar, y apartarlo de cualquier disolvente y sin interactuar con el resto de elementos que conforman el ADN y condicionan su configuración, la forma más estable de azúcares que conseguíamos era la de anillos de seis miembros”, explica Cocinero.
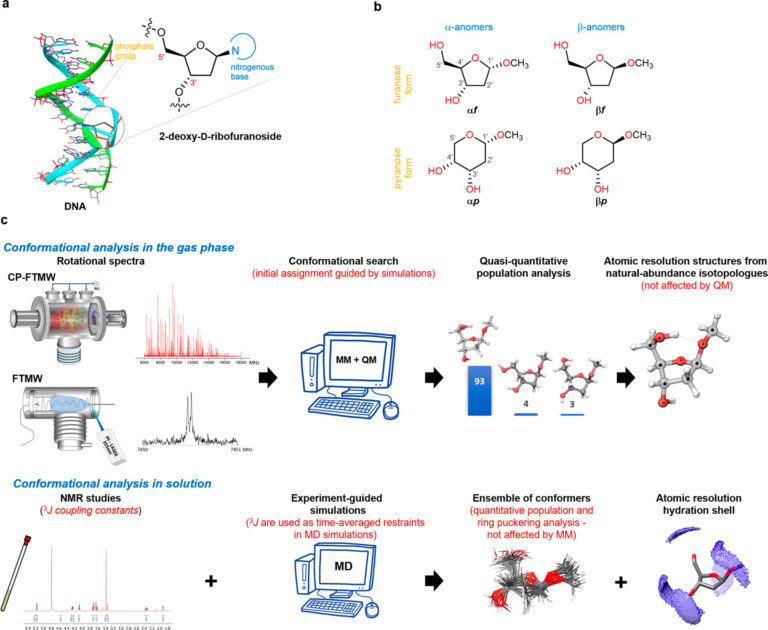
Para dar solución a esta situación, contaron con la colaboración de investigadores del Departamento de Química de la Universidad de Oxford, quienes les ayudaron en la síntesis de las cuatro formas que pueden adoptar las 2-desoxirribosas, tanto en las formas biológicas como las que no aparecen en la naturaleza, y las bloquearon, «añadiendo un grupo metilo a los azúcares para que no se interconvirtieran unas formas en otras, y poder estudiar cada una de ellas de forma individual”, detalla el investigador.
Así, han podido caracterizar la estructura de todas ellas a escala atómica, de forma aislada, y después, con ayuda de investigadores de la Universidad de La Rioja, han podido analizar cómo cambia la estructura de estas formas al entrar en contacto con el disolvente, el agua, “que se asemeja más al medio natural en el que se suelen encontrar. Hemos visto las diferencias entre unas formas y otras, y las hemos caracterizado”.
Asimismo, este análisis les ha permitido hipotetizar sobre “porqué la forma que se observa en la naturaleza es la que se observa y no otra. Según hemos visto, la forma de anillos de cinco miembros es más flexible, y la conformación que toma en la cadena del ADN favorece el enlazamiento de los consecutivos nucleótidos”, relata.
Ahora, con el desarrollo instrumental conseguido, van a abordar “el estudio de moléculas más grandes, tratando de construir sistemas que se acerquen cada vez más a las formas biológicas reales, para dar mejores respuestas. Buscamos el límite de la técnica instrumental”, concluye Emilio J. Cocinero.
Referencia:
Camilla Calabrese, Iciar Uriarte, Aran Insausti, Montserrat Vallejo-López, Francisco J. Basterretxea, Stephen A. Cochrane, Benjamin G. Davis, Francisco Corzana, and Emilio J. Cocinero (2020)Observation of the Unbiased Conformers of Putative DNA-Scaffold Ribosugars ACS Central Science 6 (2), 293-303 doi: 10.1021/acscentsci.9b01277
Edición realizada por César Tomé López a partir de materiales suministrados por UPV/EHU Komunikazioa

Ciencia inductiva y deductiva – Sicigia
[…] El azúcar del ADN caracterizado átomo a átomo por espectrometría de microondas […]