Tras el descubrimiento del ADN y su función como contenedor de las instrucciones necesarias para la construcción de un ser vivo, la genómica experimentó un crecimiento espectacular que alcanzó su punto álgido en el año 2000 con la presentación de los resultados del proyecto genoma humano. Paralelamente, se desarrollaba la transcriptómica (que nos permite analizar el mRNA presente en una célula, y en consecuencia qué genes se han expresado en un tejido) y la proteómica para el análisis de las proteínas construidas.
Pero durante la última década está ganando peso en diferentes campos el uso de una técnica que complementa la información proporcionada por las ciencias ómicas antes citadas: se trata de la metabolómica. Esta disciplina trata de realizar un análisis global de todos los metabolitos de bajo peso molecular (por debajo de 1000 Da aproximadamente) de un sistema biológico a partir de células, muestras de tejidos o fluidos corporales. ¿A qué se debe el auge de esta disciplina? ¿Qué información nos puede proporcionar un análisis detallado del metaboloma? Expliquémoslo poco a poco.
Para entender qué son los metabolitos nos vamos a sumergir en el interior de una célula. En una célula de cualquier ser vivo hay una serie de procesos que se encargan de su funcionamiento normal. Así, sabemos que el ADN contiene almacenada en genes la información necesaria para la fabricación de las proteínas del organismo y los mecanismos para regular su expresión, que estos genes se transcriben a ARN y a su vez éste se traduce finalmente a proteínas. Las proteínas, además de cumplir diversas funciones estructurales, actúan como enzimas facilitando las reacciones químicas del metabolismo celular. Estas reacciones enzimáticas se agrupan en rutas metabólicas muy complejas, en las que intervienen moléculas orgánicas como sustratos que se transforman a su vez en distintos subproductos hasta sintetizarse uno o varios productos finales. Todas estas moléculas constituyen el metaboloma de la célula.
En todo este conjunto de procesos podemos conocer con bastante precisión a la mayoría de actores que forman parten de él. Mediante la secuenciación del ADN podemos obtener la secuencia del genoma y con técnicas bioinformáticas podemos extraer de esta secuencia los genes y de estos (de una forma más o menos aproximada) el ARN y las proteínas que se construyen a partir de ellos. También existen técnicas que nos permiten analizar el ARN que se expresa en un determinado tejido y las proteínas en que se traducen. Esta relativa “facilidad” se debe a que tanto el ADN, como el ARN y las proteínas tienen una estructura polimérica con pocas unidades distintas (los cuatro nucleótidos en el caso de ADN y ARN, y los veinte aminoácidos en el caso de la proteínas) y por lo tanto podemos diseñar distintas formas de leer la secuencia de elementos individuales que las componen.
Pero con los sustratos y subproductos de las reacciones enzimáticas, los metabolitos, no ocurre lo mismo. La diversidad estructural es enorme, tan grande que no sabemos ni siquiera qué estamos buscando. Tanto es así que hoy en día, la inmensa mayoría de metabolitos de los organismos vivos nos son absolutamente desconocidos. Y aunque conozcamos con precisión los genes de una determinada especie, su expresión en las distintas células, o las proteínas que participan en una determinada ruta metabólica, ello no nos da muchas pistas de qué moléculas van a participar en las reacciones que catalicen estas enzimas o cuáles van a ser los productos finales.
¿Cómo podemos identificar los metabolitos presentes en un sistema biológico concreto (tejido, fluidos, etc)? Para ello existen distintas técnicas, pero las más utilizadas son la resonancia magnética nuclear (RMN) y la espectrometría de masas (EM) acoplada a un sistema de cromatografía líquida o gaseosa. Aunque el RMN es importante en la caracterización final de nuevos compuestos, hoy en día se prefiere el uso de EM de alta resolución por su mayor sensibilidad y requerir menor cantidad de muestra.
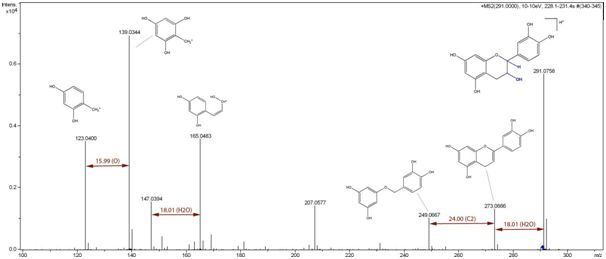
¿Y para qué nos puede interesar conocer el metaboloma? Básicamente, porque nos define el estado fenotípico de un sistema biológico en equilibrio. Y cuando este sistema recibe un estímulo externo cambia la composición del metaboloma. Por ejemplo, diferentes plantas de una misma especie vegetal, aunque tengan un ADN idéntico, pueden presentar distintos metabolomas debido a enfermedades, ataques de insectos o exposición a situaciones de estrés hídrico. El análisis de los niveles de metabolitos permiten identificar estas situaciones.
Este análisis de metabolitos podemos llevarlo a cabo de dos formas distintas, según los objetivos del estudio. Por un lado, podemos estar interesados en conocer los cambios en las concentraciones de unas moléculas concretas y previamente definidas, lo que se conoce como análisis dirigido. Pero en ocasiones no sabemos qué estamos buscando, bien porque no conocemos qué moléculas son las que cambian ante un determinado estímulo externo o enfermedad, bien porque estamos buscando nuevas especies moleculares (como ocurre en farmacología). Este tipo de estudio, mucho más complejo, se conoce como análisis no-dirigido.
El creciente aumento de investigaciones basadas en análisis metabolómicos se debe al gran número de campos en que se puede aplicar, entre los que se encuentra fisiología vegetal, nutrición, medicina, farmacología… En el resto del artículo querría exponer la utilidad de los análisis de metabolómica en la detección precoz de enfermedades y búsqueda de nuevos medicamentos.
Uno de los usos más extendidos de la metabolómica aplicada a la medicina es la búsqueda, en análisis tanto dirigidos como no dirigidos, de biomarcadores asociados a enfermedades. Los estudios llevados a cabo en este sentido utilizan como muestras tanto tejidos (como células cancerosas) como fluidos (orina, saliva, plasma sanguíneo…).
Por ejemplo, en un estudio sobre enfermedades cardiovasculares realizado con ratas publicado en la revista Nature se observó que se podía tomar el nivel sanguíneo de óxido de trimetilamina (TMAO) como medida del riesgo de diversas enfermedades, como arteriopatía periférica y coronaria, e infarto de miocardio. Posteriormente, se confirmó la validez de este indicador con una muestra de cerca de 2000 pacientes.
Otro campo médico en el que se están aplicando análisis metabolómicos es la psiquiatría, donde los diagnósticos son a veces poco claros y la necesidad de biomarcadores es más que deseable. También se ha estudiado mediante metabolómica la respuesta de los pacientes a la medicación en diversos trastornos psiquiátricos, como esquizofrenia y depresión. En estas enfermedades, aunque también se han buscado variaciones en el metaboloma de la sangre, parece que resulta más conveniente analizar el líquido cefalorraquídeo.
Un claro ejemplo en el que sería deseable un diagnóstico precoz es el de los distintos tipos de cáncer, y en ese sentido existe una gran cantidad de investigación que se basa en análisis metabolómicos. Es bien conocido que el metabolismo de las células cancerosas es distinto al de las células sanas, por lo que la búsqueda de biomarcadores que diferencie ambas siempre ha sido un objetivo en el diagnóstico de cáncer. En concreto, sería deseable una detección precoz utilizando métodos poco invasivos (que no impliquen la extracción de una muestra de tejido canceroso). Por ejemplo, en un estudio del suero sanguíneo de pacientes con cáncer de páncreas (uno de los tumores con un diagnóstico precoz más complejo) un grupo de investigadores detectó que 18 de los 60 metabolitos analizados variaba de forma significativa entre los sujetos sanos y los pacientes, incluso en las primeras fases del cáncer. Este resultado podría utilizarse para la detección precoz del cáncer de páncreas. En el cáncer de mama, uno de los tipos de tumores más estudiados, existen un gran número de investigaciones basadas en análisis metabolómicos que apuntan a que las variaciones de concentración de distintos tipos de lípidos podrían ser un indicador de la fase de desarrollo de este tipo de cáncer.
Otro campo de interés de la metabolómica es la búsqueda de nuevos fármacos. Un gran número de los fármacos que usamos hoy en día son metabolitos de plantas o microorganismos, o derivados químicos de estos— de hecho, una tercera parte de los aprobados por la FDA entre 1981 y 2010. Se estima que las bacterias producen del orden de cientos de metabolitos, mientras que las plantas generarían del orden de miles. Son particularmente interesantes los metabolitos secundarios de las plantas, que éstas fabrican como mecanismo de autodefensa frente a depredadores o como adaptación al medio. Sin embargo, aún hoy en día no conocemos la mayor parte de los metabolitos presentes en los seres vivos.
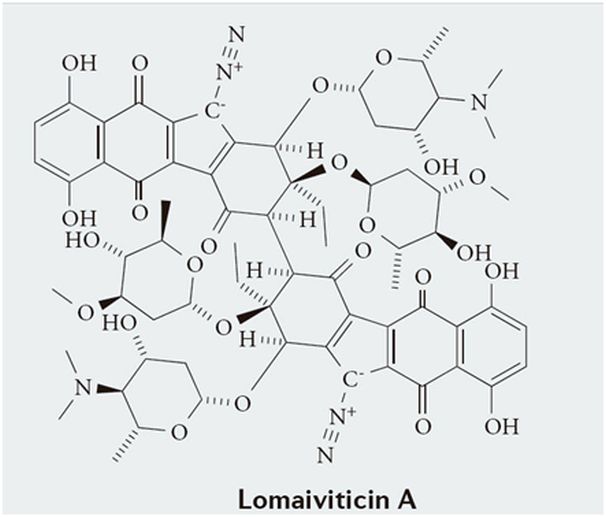
Durante los últimos años la industria farmacéutica había abandonado la búsqueda de nuevos fármacos basados en productos naturales, confiando en técnicas de química combinatoria. Pero los avances en técnicas de análisis químico han vuelto a situar en el foco a este tipo de sustancias. En este caso también estaríamos interesados en un análisis metabolómico no dirigido, porque de hecho estamos buscando nuevas moléculas. Obviamente, la elucidación estructural de nuevos compuestos a partir de los espectros de masas resulta aún más compleja por no existir referencias en las bases de datos moleculares o de espectros.
En esta breve introducción a la metabolómica no he podido hablar de sus usos en otros campos, como la aplicación en tecnología de alimentos, control de calidad del medio ambiente, fisiología animal… Tampoco de las dificultades con las que se encuentran los investigadores a la hora de elucidar las estructuras moleculares a partir de espectros de masas y la ayuda que les puede prestar la bioinformática. Aunque de esto último espero escribir en breve.
Este post ha sido realizado por Guillermo Peris (@Waltzing_piglet) y es una colaboración de Naukas con la Cátedra de Cultura Científica de la UPV/EHU.
Referencias
- Putri et al. Current metabolomics: Practical applications. Journal of Bioscience and Bioengineering vol. 115 No. 6, 579e589 (2013) doi:10.1016/j.jbiosc.2012.12.007
- Alan L. Harvey, RuAngelie Edrada-Ebel & Ronald J. Quinn. The re-emergence of natural products for drug discovery in the genomics era. Nature Reviews Drug Discovery 14, 111–129 (2015) doi:10.1038/nrd4510
- Emily G. Armitage, Coral Barbas. Metabolomics in cancer biomarker discovery: Current trends and future perspectives. Journal of Pharmaceutical and Biomedical Analysis 87, 1-11 (2014) doi:10.1016/j.jpba.2013.08.041

Próxima estación: metaboló…
[…] Tras el descubrimiento del ADN y su función como contenedor de las instrucciones necesarias para la construcción de un ser vivo, la genómica experimentó un crecimiento espectacular que alcanzó su punto álgido en el año 2000 con la… […]
Próxima estación: metabolómica
[…] Próxima estación: metabolómica […]
Hitos en la red #86 – Softayuda
[…] colores de Plutón en alta resolución de Dani, o los entresijos de la fisiología/bioquímica en Próxima estación: metabolómica por Guillermo Peris. Y ya que estamos con la estética: Qué sería del arte sin la ciencia y […]
Próxima estación: metaboló…
[…] Tras el descubrimiento del ADN y su función como contenedor de las instrucciones necesarias para la construcción de un ser vivo, la genómica experimentó un crecimiento espectacular que alcanzó su punto álgido en el año 2000… […]