La Facultad de Ciencias de Bilbao comenzó su andadura en el curso 1968/69. 50 años después la Facultad de Ciencia y Tecnología de la UPV/EHU celebrará dicho acontecimiento dando a conocer el impacto que la Facultad ha tenido en nuestra sociedad. Durante las próximas semanas en el Cuaderno de Cultura Científica y en Zientzia Kaiera se publicarán regularmente artículos que narren algunas de las contribuciones más significativas realizadas a lo largo de estas cinco décadas. Comenzamos con la serie “Espectrometría de masas”, técnica analítica que supone el ejemplo perfecto del incesante avance de la Ciencia y la Tecnología.
Durante la última mitad del siglo pasado se vivió una gran revolución en la biología. Importantes avances tecnológicos y científicos (con el descubrimiento de la doble hélice de ADN como el ejemplo más conocido) hicieron del reduccionismo la máxima de esta ciencia, descomponiendo organismos complejos para estudiar sus partes constituyentes aisladamente, lo que dio lugar a una gran expansión de la biología molecular. Sin embargo, en los últimos años la biología está viviendo un nuevo cambio de rumbo, en parte experimental y en parte filosófico, hacia una visión holística o integral del sistema, tomando el conjunto de componentes individuales y su interacción como objeto de estudio. En palabras de Aristóteles, el todo es más que la suma de las partes. Estas diferencias entre la visión reduccionista del siglo pasado y la actual biología de sistemas pueden compararse con la leyenda india de los sabios ciegos y el elefante, en la que se representan las limitaciones de una visión individual, o reducida, en la aproximación a un problema desconocido.

En esta leyenda seis sabios ciegos que nunca habían visto un elefante deciden buscar uno y tocarlo para poder hacerse una imagen mental del animal. El primero de ellos al acercarse tropieza y cae de bruces contra el costado del animal. De esta experiencia deduce que un elefante debe ser algo parecido a una pared de barro. El segundo sabio toca el colmillo del elefante y de su forma redonda y afilada infiere que un elefante es algo parecido a una lanza. De un modo similar, los otros cuatro sabios tocando la trompa, la cola, una pata y una oreja, deciden individualmente que ese animal, para ellos hasta ahora desconocido, es similar a una serpiente, una cuerda, el tronco de un árbol o un abanico, respectivamente. Al discutir sus descubrimientos no llegan a un acuerdo sobre la forma del elefante, ya que, como dice John Godfrey Saxe en su poema sobre esta leyenda “aunque todos estaban parcialmente en lo cierto, todos estaban equivocados”. Del mismo modo, pretender comprender la totalidad de sistemas biológicos complejos basándose solamente en observaciones parciales puede llevarnos a conclusiones que sean, si no incorrectas, incompletas.
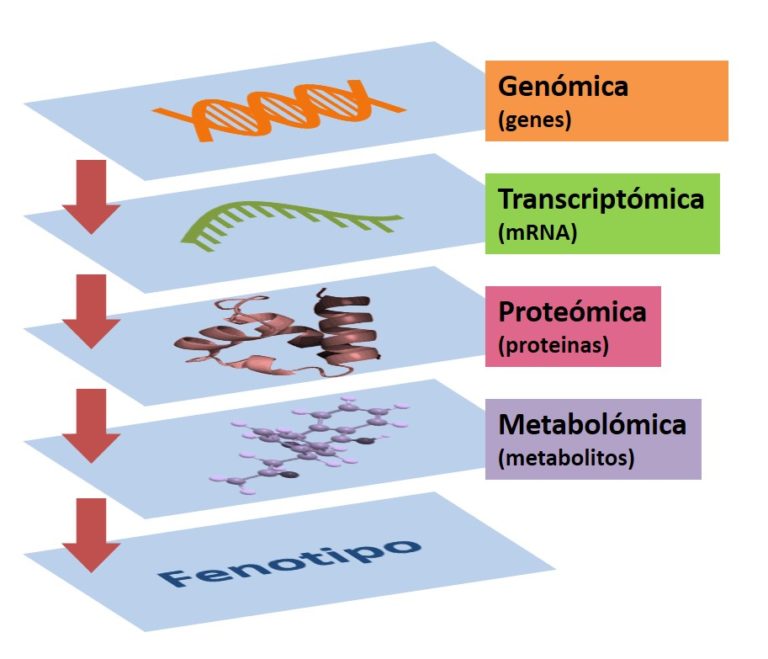
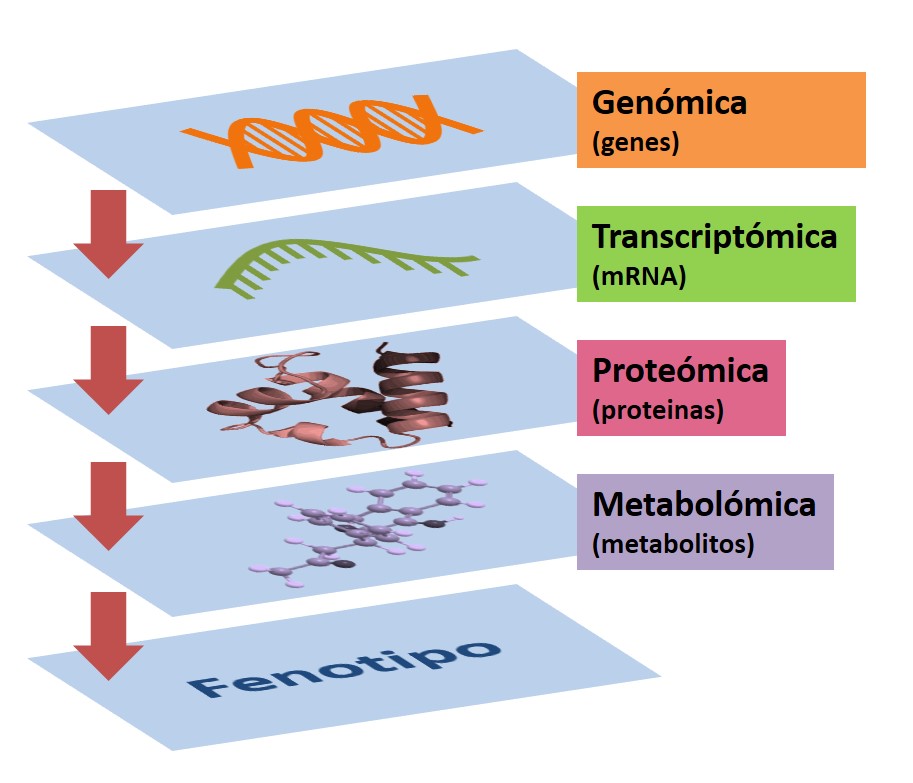
En la rápida expansión de la biología de sistemas ha tenido gran influencia la aparición de las tecnologías ómicas. Entre ellas la más conocida hasta ahora ha sido la genómica, con el Proyecto Genoma Humano como gran desafío tecnológico de finales del siglo XX y comienzos del XXI. Este proyecto, en el cual se pretendía la secuenciación total del genoma humano, fue completado en 2003, dos años antes de lo previsto, ofreciendo a la comunidad científica información detallada sobre la estructura, organización y función del conjunto completo de genes humanos: el genotipo.
El genotipo es, en gran parte, responsable del estado final de un organismo. No obstante, el fenotipo, es decir, la descripción del total de las características físicas de un sistema biológico incluyendo su morfología, desarrollo y metabolismo, está también fuertemente influenciado por factores ambientales. La metabolómica se considera la última disciplina en la cascada de las ómicas, la más cercana al fenotipo, y por tanto la más representativa del estado del organismo en un momento puntual.
La importancia del estudio de los metabolitos en fluidos biológicos se remonta al 1500-2000 antes de la era actual, cuando tanto en la medicina tradicional china como en la Ayurveda practicada en la India se utilizaban insectos para detectar niveles altos de glucosa en la orina de los pacientes. Los primeros experimentos en metabolómica pueden considerarse los del doblemente laureado por los premios Nobel Linus Pauling, que en 1971 analizó alrededor de 250 metabolitos en muestras de aliento y vapor de orina, dando lugar a la idea de que a partir de un patrón generado por un elevado número de metabolitos en un fluido biológico se puede recoger información sobre el estado de un sistema biológico complejo. Sin embargo, el gran boom de la metabolómica tuvo lugar a final de los años 90. Fue en 1999 cuando Nicholson acuñó el término metabonómica para describir “la medida cuantitativa de la respuesta dinámica y multiparamétrica de los sistemas vivos a estímulos patofisiológicos o genéticos”, es decir, cuantificar a través del estudio del conjunto de metabolitos el estado de un ser vivo debido a su información genética o a un cambio externo. Desde entonces el uso de la metabolómica ha crecido exponencialmente (llegando a más de 4000 publicaciones con el término metabolomics en PubMed en 2018) y se aplica en áreas tan diversas como el estudio de enfermedades, el desarrollo de fármacos, la ciencia forense, el análisis medioambiental, la nutrición o la toxicología, entre otros.
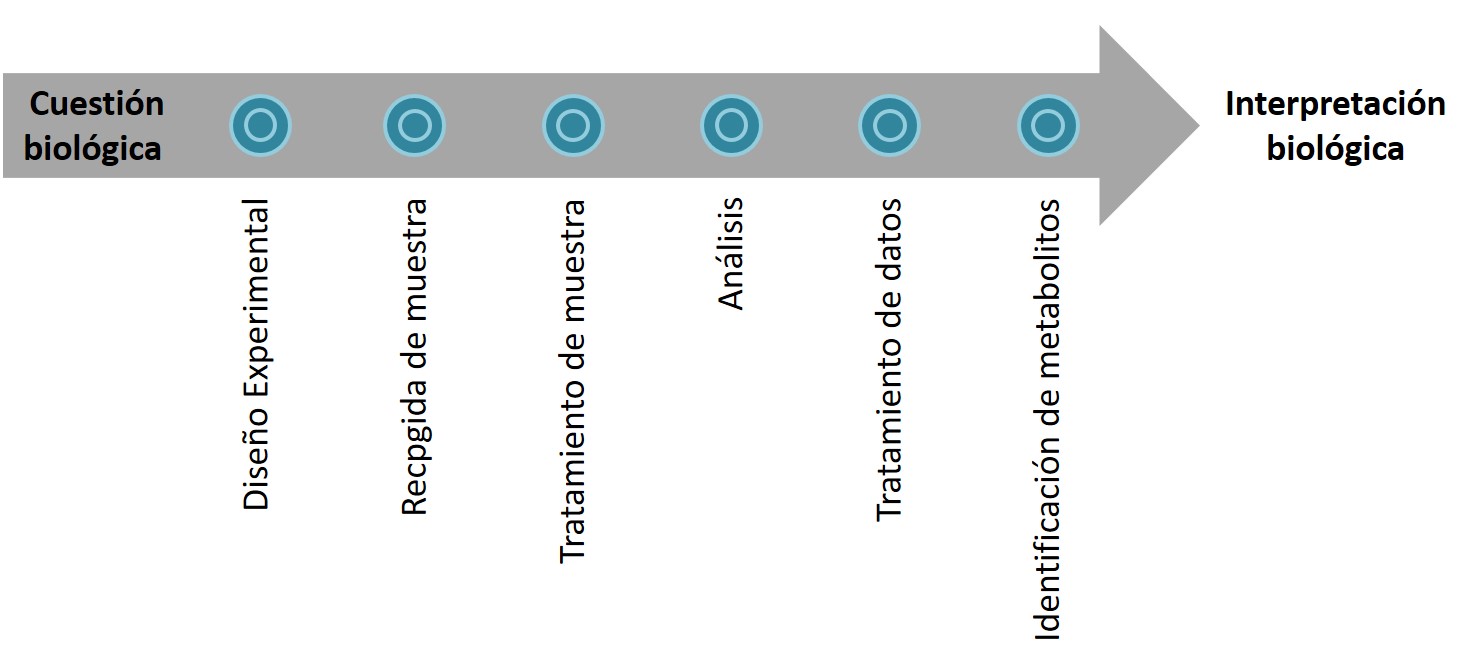
La metabolómica es una disciplina amplia y compleja, que requiere de diversos pasos para llegar desde la cuestión biológica, es decir, el planteamiento del problema (por ejemplo, qué diferencia un individuo sano de uno enfermo, qué cambios provoca en el metabolismo un cambio de dieta, qué relación hay entre el desarrollo de un niño y el efecto en él de un fármaco, o cómo afecta a un sistema un compuesto tóxico, entre muchas otras) a la interpretación de los resultados. El primero, y uno de los más importantes, es el diseño del estudio. Éste debe realizarse con la colaboración de todas las personas implicadas a lo largo de todos los pasos, desde la toma de muestra al análisis estadístico y la interpretación biológica. Como bien dijo Sir Ronald Aylmer Fisher: “consultar al especialista en estadística después de realizar el experimento es como pedirle que realice un análisis post-mortem: posiblemente pueda decir de qué murió el experimento”. Antes de comenzar el experimento deben definirse perfectamente el resto de pasos (toma y tratamiento de muestra, análisis de las muestras, tratamiento y procesado de los datos) para llegar a la interpretación de resultados.
El objetivo de la metabolómica es estudiar el metaboloma completo, aunque en contraste con el genoma o el proteoma, el metaboloma no es fácilmente definible. En ocasiones se define como el conjunto de metabolitos sintetizados por un sistema biológico, siendo este sistema un organismo, órgano, tejido, fluido, célula… Pero entre los metabolitos presentes en el cuerpo humano no encontramos solo compuestos endógenos, sino que también se encuentran los productos de aquello que ingerimos o con lo que estamos en contacto, es decir, los metabolitos exógenos.
Los metabolitos, tanto endógenos como exógenos, constituyen una familia muy heterogénea de moléculas, con muy diversas estructuras, propiedades físico-químicas y concentraciones. Esta heterogeneidad hace que, por el momento, nos sea imposible medir simultáneamente todo el metaboloma usando una única técnica. Es por ello que para poder cubrir el máximo rango posible del metaboloma debemos usar diferentes plataformas analíticas. Especialmente durante los inicios de la metabolómica primaba el uso de la resonancia magnética nuclear (RMN). Sin embargo, la metabolómica basada en la espectrometría de masas (MS) ha ido ganando en popularidad con el tiempo. El desarrollo de instrumentos de alta resolución como la resonancia ciclotrónica con transformada de Fourier (FTICR), el Orbitrap o el tiempo de vuelo (TOF), junto a los bajos límites de detección y la rapidez del análisis han hecho que actualmente la MS sea la técnica elegida en la mayoría de estudios metabolómicos.
Aunque existen algunos estudios en los que la muestra se introduce al MS por infusión directa (DI-MS), lo más común es que se acople al espectrómetro una técnica de separación que ayude a reducir la complejidad de los espectros y a disminuir la supresión iónica debida a la competición por la ionización de las miles de moléculas distintas presentes simultáneamente en la muestra. Dependiendo de los analitos de interés usamos distintas técnicas. Para estudiar compuestos volátiles la técnica de elección es la cromatografía de gases acoplada a MS (GC-MS). Ésta fue la técnica más empleada en los inicios de la metabolómica basada en MS, especialmente aplicada al estudio de las plantas, pero tiene el inconveniente de que para analizar metabolitos no volátiles debemos derivatizarlos, lo que requiere un tratamiento de muestra complejo y tedioso. En el caso de metabolitos cargados, es común el uso de la electroforesis capilar (CE-MS). Hoy en día la cromatografía líquida acoplada a la MS (LC-MS) es sin duda la técnica de primera elección, que permite el estudio de compuestos tanto polares como apolares. Dado que ninguna de las técnicas es capaz por si sola de analizar el metaboloma completo, el uso de técnicas complementarias es altamente recomendable.
Los análisis metabolómicos producen gran cantidad de datos que necesitan software y metodologías específicas para su tratamiento. Del sistema de LC-MS obtenemos un set de datos tridimensional que debemos simplificar para poder trabajar con él, de modo que obtengamos una matriz bidimensional con una lista de “features” y sus intensidades. Aún simplificada, se trata de una matriz compuesta por miles de estos features hasta en cientos de muestras que debe someterse a varios tratamientos más (normalización, transformación, centrado, escalado…) hasta llegar a un set de datos adecuado para ser estudiado mediante estadística multivariante. El objetivo de este tratamiento de datos es obtener una lista de features responsables de las diferencias entre los distintos grupos estudiados (enfermos vs sanos, jóvenes vs ancianos, tratados vs no tratados…), seleccionados como posibles biomarcadores que nos ayuden a responder la hipótesis planteada.

El último paso antes de la interpretación biológica de los resultados es la identificación de los features seleccionados, sirviéndose de la información que nos da el instrumento de LC-MS (tiempo de retención, masa exacta, espectro de fragmentación) para buscar los nombres de los metabolitos seleccionados como biomarcadores. Dada la gran cantidad y diversidad química de metabolitos existentes la identificación es aún hoy en día el cuello de botella de la metabolómica basada en LC-MS. Existe un gran número de metabolitos en el cuerpo humano que no han sido aún identificados, a pesar de los grandes esfuerzos realizados por la comunidad científica. Sin embargo, bases de datos como METLIN, Lipid Bank, KEGG, Lipid Maps o HMDB continuamente registran nuevas entradas que amplían los metabolitos disponibles. En ocasiones la identificación de estos metabolitos responde por sí misma a la cuestión biológica planteada, pero generalmente abre nuevas vías de investigación hacia la solución del problema. Por ejemplo, si encontramos cambios en la concentración de varios compuestos de una misma ruta metabólica en individuos enfermos respecto a controles sanos, esto nos da una idea de la alteración de esta ruta, de modo que pueda ser estudiada de un modo más dirigido.
Un ejemplo concreto de aplicación de la metabolómica es el estudio de la toxicidad de materiales derivados del grafeno. El grafeno es uno de los grandes descubrimientos de la actualidad: una red de atomos de carbono bidimensional (con el espesor de un átomo) con unas prometedoras propiedades en cuanto a dureza, flexibilidad o conductividad entre muchas otras, que lo hacen muy atractivo para numerosas aplicaciones. Entre ellas se estudia su uso en biosensores o como conductor de fármacos. Sin embargo, por ser un compuesto apenas conocido el efecto que puede producir su contacto con las células es totalmente desconocido. En este caso es de gran utilidad el uso de las tecnologías ómicas, que nos permite buscar diferencias entre células expuestas a estos compuestos frente a células control, explorando de manera no dirigida, sin una hipótesis previa, los cambios producidos en el sistema.

Los estudios metabolómicos son complejos y requieren un gran trabajo y esfuerzo, pero su gran atractivo reside en su utilidad en los casos en los que el problema planteado es apenas conocido y no existe información anterior que permita hacer un análisis dirigido. Si, como hemos dicho, son miles los metabolitos presentes en el cuerpo humano, estudiarlos dirigidamente pretendiendo encontrar el biomarcador justo puede considerarse una tarea prácticamente imposible, como encontrar una aguja en un pajar, pero sin saber que lo que buscamos es una aguja.
Sobre la autora: María Encarnación Blanco se doctoró en la Facultad de Ciencia y Tecnología de la UPV/EHU y actualmente es investigadora en el Istituto Italiano di Tecnologia. Trabaja en la aplicación de la metabolómica en el estudio de la toxicidad de compuestos de grafeno en el cerebro dentro del proyecto europeo EU Graphene Flagship Project Horizon 2020 Research and Innovation Programme (Grant agreement no. 785219).