La Facultad de Ciencias de Bilbao comenzó su andadura en el curso 1968/69. 50 años después la Facultad de Ciencia y Tecnología de la UPV/EHU celebrará dicho acontecimiento dando a conocer el impacto que la Facultad ha tenido en nuestra sociedad. Durante las próximas semanas en el Cuaderno de Cultura Científica y en Zientzia Kaiera se publicarán regularmente artículos que narren algunas de las contribuciones más significativas realizadas a lo largo de estas cinco décadas. Comenzamos con la serie “Espectrometría de masas”, técnica analítica que supone el ejemplo perfecto del incesante avance de la Ciencia y la Tecnología.
La espectrometría de masas es una poderosa técnica analítica que permite determinar la identidad de las moléculas a partir del dato de su masa. Para la aplicación de la técnica de espectrometría de masas, la muestra, independientemente del estado en que se encuentre, debe ser transferida intacta y cargada eléctricamente a fase gas. Por supuesto, esta definición es una supersimplificación de los principios físico-químicos que subyacen a la técnica. Sin embargo, de ella se desprenden varios datos importantes: la técnica permite analizar muestras muy complejas sin marcado previo (es decir, sin necesidad de marcar las moléculas que se quieren detectar). Estas dos características, junto con la de su enorme precisión, sensibilidad y velocidad, la hacen especialmente útil para el análisis de muestras biológicas. Sin embargo, se necesitó del desarrollo de las denominadas técnicas de ionización blanda, para poder transferir las moléculas biológicas intactas a fase gas.
La técnica de MALDI (Matrix Assisted Laser Desoption/Ionization, Figura 1) es una de las más utilizadas para la introducción de moléculas termolábiles en un espectrómetro de masas. Comienza depositando una porción representativa de la muestra biológica en una placa de acero y recubriéndola con una sustancia orgánica (la matriz), que absorbe la luz en la zona de emisión de los láseres comerciales (normalmente en el UV, en torno a los 335-350 nm). Las moléculas biológicas son, normalmente, transparentes a esta radiación, por lo que no se ven afectadas por el láser. Sin embargo, la matriz sí absorbe la radiación láser, se fragmenta, y libera el analito ionizado en fase gas.
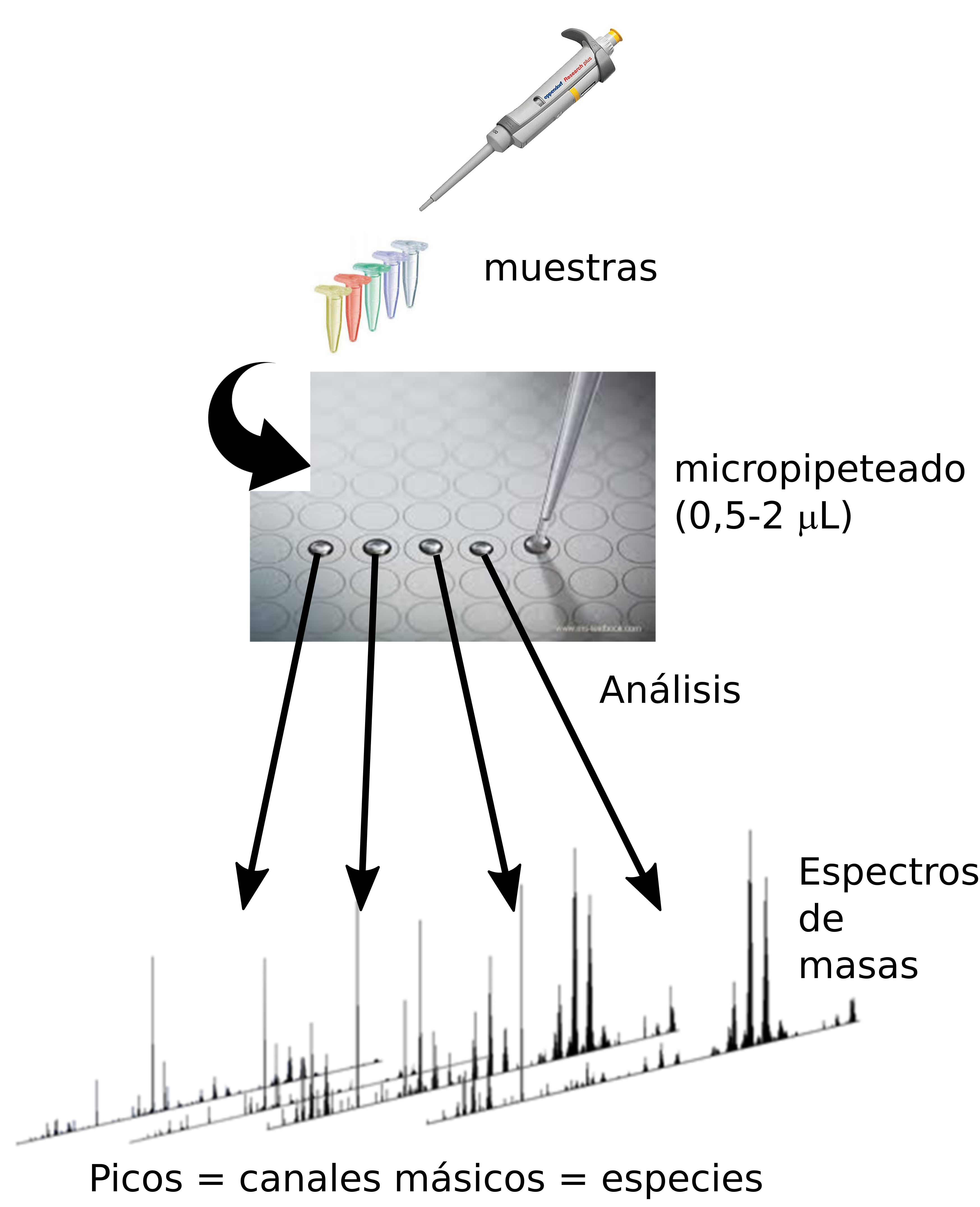
En los años 90, surge un refinamiento de esta técnica: la imagen por espectrometría de masas, que permite la exploración directa de tejidos (Figura 2). Efectivamente, la aplicación de la metodología MALDI no tiene por qué restringirse a muestras sólidas, sino que se puede aplicar directamente sobre secciones de tejido congelado en fresco. De este modo, se obtienen los mapas de distribución de todas las moléculas biológicas (detectables) a lo largo del tejido. La técnica de MALDI-IMS (MALDI-Imaging Mass Spectrometry), es capaz no sólo de dar la identificación de cientos de biomoléculas en un solo experimento, sino que aporta su localización dentro del tejido (información espacial), permitiendo asociar los cambios a estructuras histológicas o incluso a células individuales.
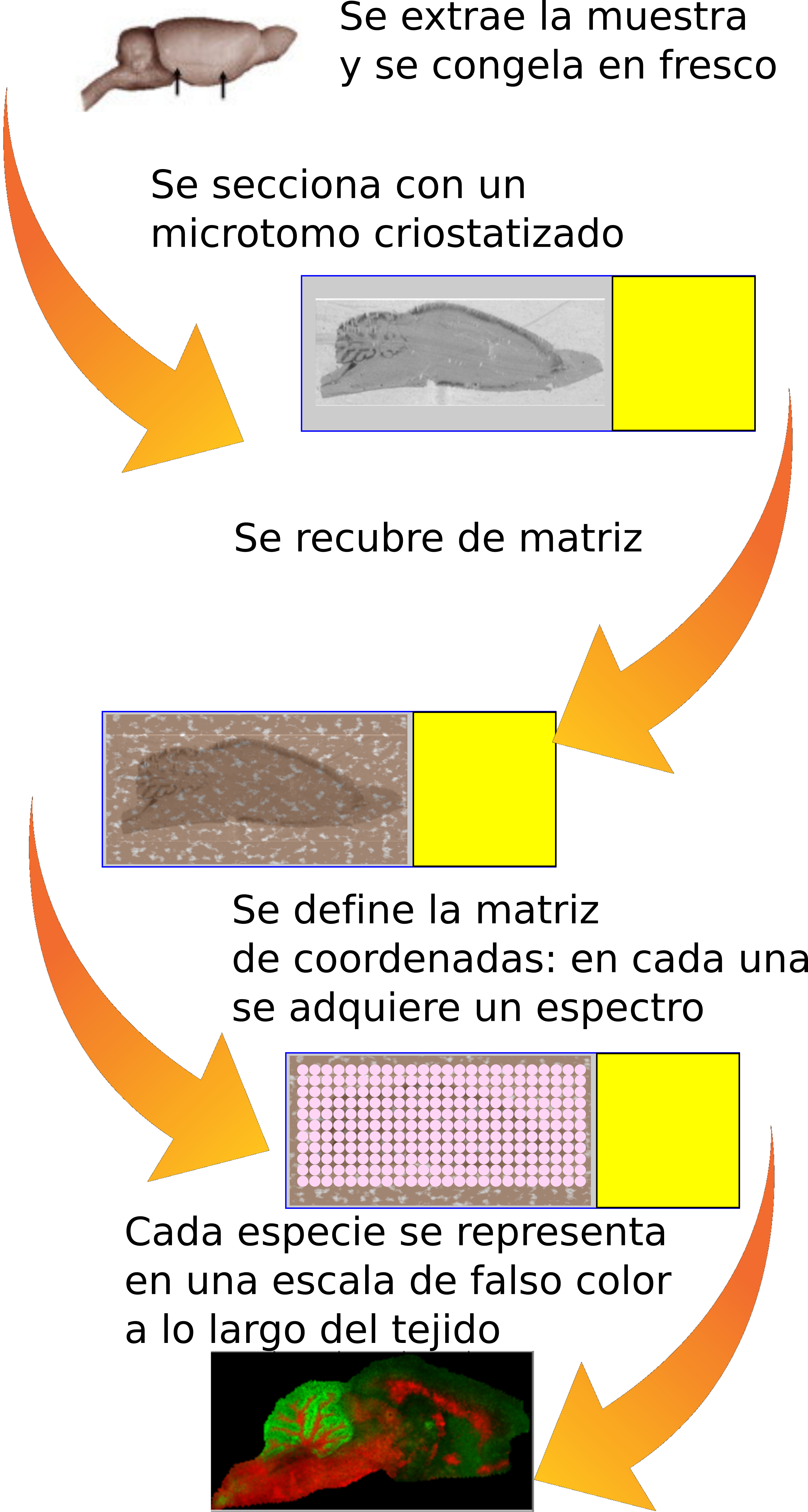
Estudio de células de colon
En metabolómica la información espacial es de enorme relevancia, ya que cada tipo celular presenta un perfil metabólico propio, que cambia con el ciclo celular. Por ejemplo, la técnica de IMS ha sido de gran utilidad para estudiar la maduración de células en el colón, la última parte del sistema digestivo. La pared de este órgano (Figura 3), está recubierta de unas invaginaciones, denominadas criptas, que están formadas por una única capa de células epiteliales (vamos, de piel). Como todos los epitelios, el del colon también sufre un desgaste, por lo que las células deben reemplazarse continuamente. En el fondo de las criptas, existe un nicho de células madre en constante división, que son las encargadas de abastecer de nuevas células a la cripta. La presión de estas nuevas células, empuja hacia arriba a la anteriores, que van madurando para adoptar papeles especializados, hasta que, al llegar a la parte exterior, descaman.
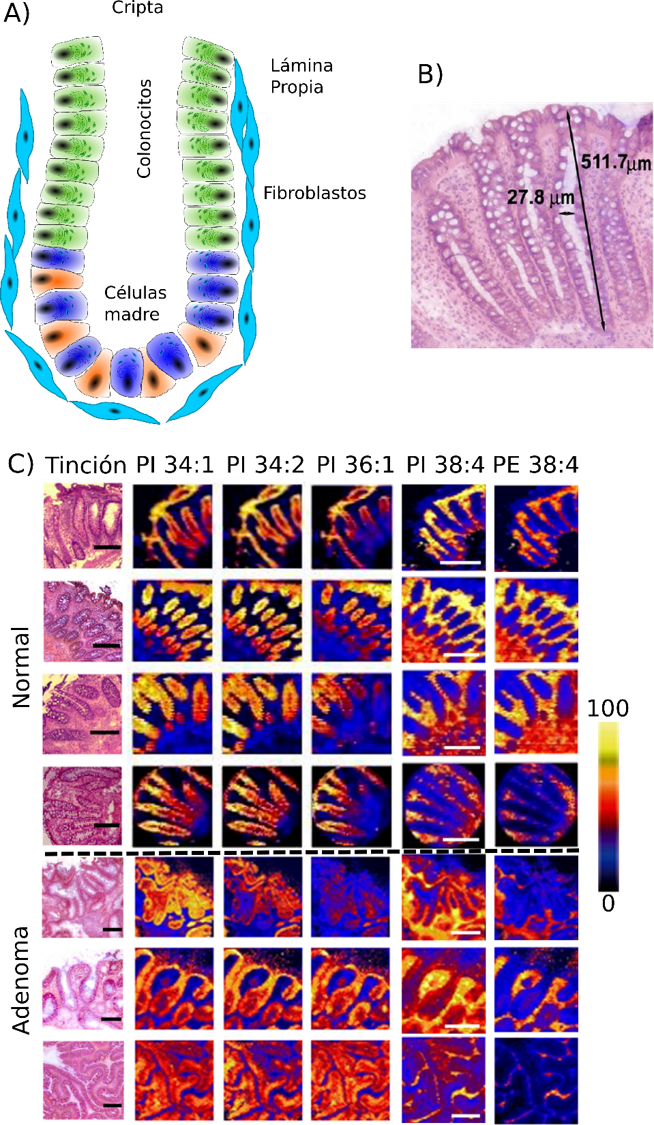
Gracias a nuestros experimentos de MALDI-IMS en colaboración con el grupo de la Dra. Barceló-Coblijn (IdisPa, Palma), hemos demostrado que todo el proceso de división y maduración de las células, está acompañado por un cambio en su perfil lipídico o lipidoma (el conjunto de los lípidos que contiene la célula): a medida que el colonocito va madurando, las especies lipídicas que contienen ácido araquidónico (un ácido graso con funciones señalizadoras), van reemplazándolo por otros ácidos grasos de cadena más corta y con menor número de insaturaciones. En resumen, los lípidos están tan estrictamente regulados, que se puede deducir a qué altura de la cripta se encuentra un colonocito, simplemente observando su perfil lipídico. Más aún, las imágenes nos permitieron determinar que este cambio en el lipidoma se da en la parte central y terminal del colonocito, mientras que el núcleo del mismo no presenta cambio alguno en su lipidoma con la maduración.
Las criptas colónicas se encuentran rodeadas de otro tejido denominado lámina propia, que le da soporte y que está altamente infiltrado por células del sistema inmune, siempre vigilantes ante posibles invasiones bacterianas. No debemos olvidar que, del otro lado del epitelio, existe una inmensa población bacteriana que nos ayuda a procesar los alimentos y a aprovechar los nutrientes. Mientras las bacterias estén del lado correcto, son beneficiosas. Lo impresionante de la técnica de MALDI-IMS es que nos permitió demostrar que las células de la lámina propia siguen un patrón completamente diferente: presentan una enorme cantidad de ácido araquidónico, que está relacionado con la inflamación, y que éste es más abundante cerca de la parte luminal del colon. Es decir, células contiguas, colonocito y fibroblasto, presentan lipidomas completamente distintos y regulan su expresión lipídica de manera independiente. Es decir, cada célula tiene una composición de lípidos propia y exclusiva, que permite su identificación
Una aplicación directa y evidente de todo este conocimiento es la detección temprana del cáncer de colon. Si el perfil lipídico de una célula está tan estrictamente regulado, la alteración metabólica que el proceso de malignización produce, debería tener un impacto dramático en el perfil lipídico celular. De hecho, nuestros resultados demuestran que el tejido alterado de colon, presenta alteraciones significativas, tanto morfológicas como metabólicas. La Figura 3 muestra imágenes histológicas de una sección de biopsia de colon neoplásico. Claramente, las criptas han perdido su morfología típica, debido a que las células madre se reproducen descontroladamente, produciendo demasiados colonocitos, que además no maduran. Tal y como esperábamos, la huella lipídica de estos colonocitos coincide con la huella lipídica de las células madre en el fondo de la cripta, demostrando desde un punto de vista molecular, que son colonocitos inmaduros que se siguen reproduciendo y que no llegan a diferenciarse. Mientras tanto, las células de la lámina propia, siguen ajenas al proceso de malignización y siguen presentando un perfil lipídico similar al que presentan en tejido sano.
Este ejemplo demuestra la importancia de la utilización de técnicas con localización espacial para el estudio del metaboloma, y más concretamente, del lipidoma, ya que los cambios se producen a nivel celular. Sin la resolución espacial, los cambios producidos en los colonocitos, quedarían diluidos entre el lipidoma del resto de las células.
Estudio del cerebro
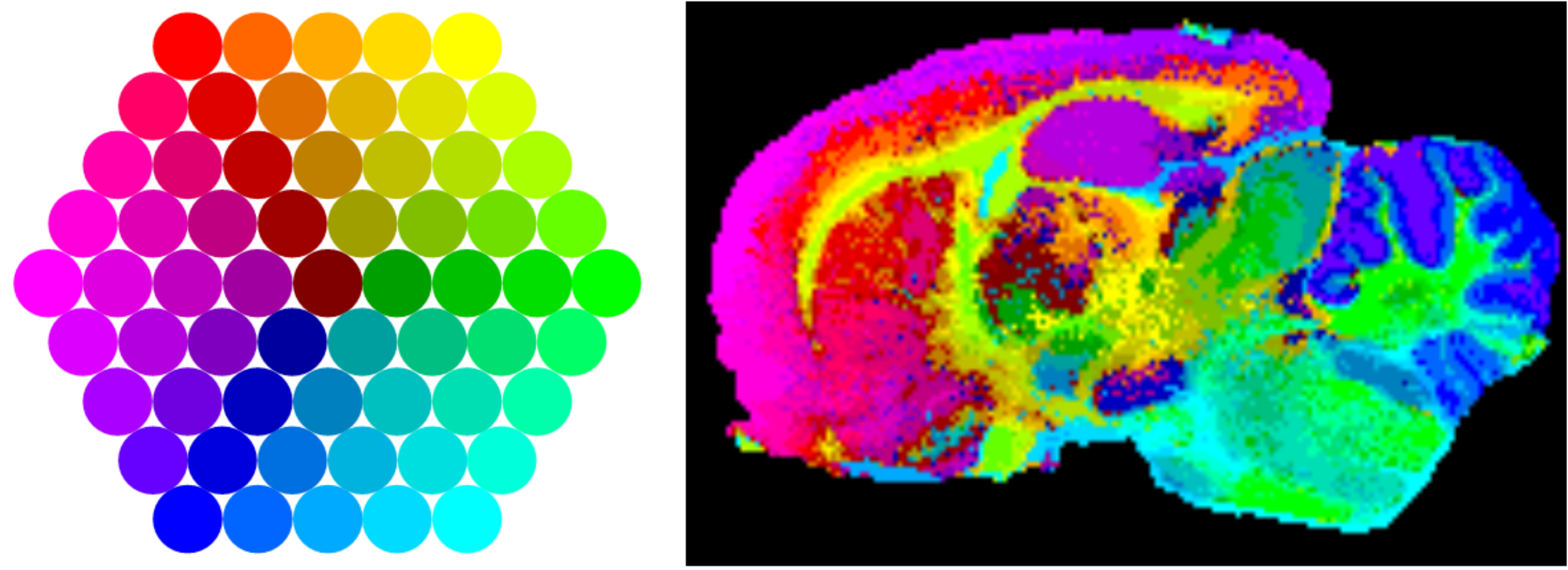
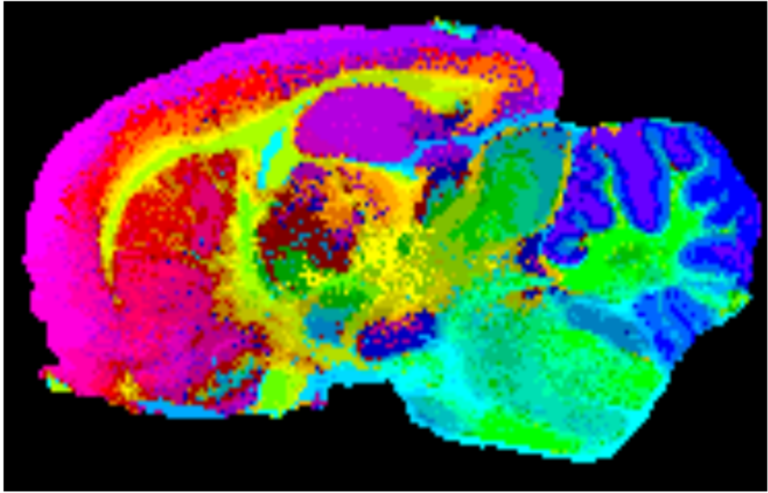
La aplicación de la técnica de MALDI-IMS está suponiendo una revolución no sólo en el campo de la lipidómica, sino también en proteómica y en campos más aplicados como la anatomía patológica. Efectivamente, del mismo modo que en los ejemplos expuestos en este artículo se presentan mapas de distribución de lípidos, también es posible la detección de proteínas y péptidos directamente en tejido y realizar las imágenes correspondientes. Realmente, la técnica es aplicable a cualquier molécula detectable por MALDI y permite al patólogo ver la histología de un tejido, desde un punto de vista molecular. Como cada tipo de tejido o incluso de célula tiene su perfil lipidómico/proteómico propio, se pueden utilizar herramientas de análisis estadístico para visualizar los píxeles en base a su huella molecular, como se muestra en la Figura 4, donde se muestra el resultado de un experimento de MALDI-IMS sobre una sección sagital (a lo largo, desde el bulbo olfatorio hasta el cerebelo) de cerebro de rata: los espectros de cada pixel se han analizado mediante un algoritmo de análisis estadístico y se han proyectado después sobre una red neuronal, utilizando el patrón de colores mostrado en la imagen: aquellos píxeles con un perfil metabólico más parecido, presentan colores más próximos en la escala. Se observa que la imagen resultante reproduce fielmente la histología del tejido. Cualquier alteración del metabolismo de un tipo de célula, resultaría en la alteración del patrón de colores, permitiendo fácilmente su identificación.
En resumen, la técnica de MALDI-IMS permite añadir una dimensión extra al análisis de muestras biológicas: la localización espacial dentro de un tejido de los metabolitos y proteínas que lo componen, abriendo un abanico de futuras aplicaciones en el estudio de las enfermedades metabólicas.
Sobre el autor: José A. Fernández es investigador en el Departamento de Química Física de la Facultad de Ciencia y Tecnología de la UPV/EHU

Proteómica: cuando las proteínas y la espectrometría de masas van de la mano – Fluceando
[…] por desorción láser asistida por matriz, (matrix assisted laser desorption ionization) (MALDI). En ESI, se aplica un alto voltaje a la solución que contiene el analito a su paso por una fina […]