Guillermo Peris
El pasado mes de diciembre se publicó un artículo de investigación que dio lugar a una gran polémica por sus conclusiones. En él se afirmaba haber encontrado evidencias de que el SARS-CoV-2 es capaz de integrarse en el genoma humano de los afectados por COVID-19. Según sus autores, esto explicaría que algunos infectados por este virus den positivo meses después de haber pasado la enfermedad, ya que las células de estos individuos producirían fragmentos del virus que serían detectados por una RT-PCR. Este artículo fue compartido en biorXiv, una plataforma en la que se publican investigaciones antes de pasar por un proceso de revisión por pares previo a la publicación en una revista científica. Como era de esperar, el artículo dio alas a los grupos antivacunas que lo usaron para denunciar que esta integración también podría darse con vacunas de ARN mensajero.
A principios de mayo de 2021 el artículo fue publicado online en la revista PNAS (Proceedings of the National Academy of Sciences of the United States of America, factor de impacto 9.4 en 2019) por un procedimiento no menos polémico: los socios de la National Academy of Sciences pueden enviar hasta dos artículos al año como contributed authors, en los que ellos eligen previamente a los dos revisores que evaluarán su trabajo. En este caso, el autor principal Rudolf Jaenisch siguió este camino para publicar su estudio. Además, en esta versión final intentó responder a las críticas que tuvo el artículo original en bioRxiv (y que fue rechazado por otra revista). Sin querer entrar a valorar las posibles implicaciones de esta forma sesgadas de revisión por pares (para lo que te aconsejo leer este artículo de Francis Villatoro), en este artículo quiero analizar únicamente las evidencias presentadas por los investigadores con las que pretenden respaldar su hipótesis de que el SARS-CoV-2 podría integrarse en los genomas celulares de los infectados. Para ello intentaré describir de la forma más sencilla posible los experimentos realizados y si realmente de los resultados se pueden inferir las conclusiones de este estudio.
LINE-1, el presunto compinche de SARS-CoV-2
El virus SARS-CoV-2 pertenece a la familia de los coronavirus y almacena su material genético en forma de ARN de una sola hebra (monocatenario) en el interior de una membrana formada por distintas proteínas, entre ellas la conocida como espícula o proteína S en la que se basan las vacunas de Pfizer y Moderna.
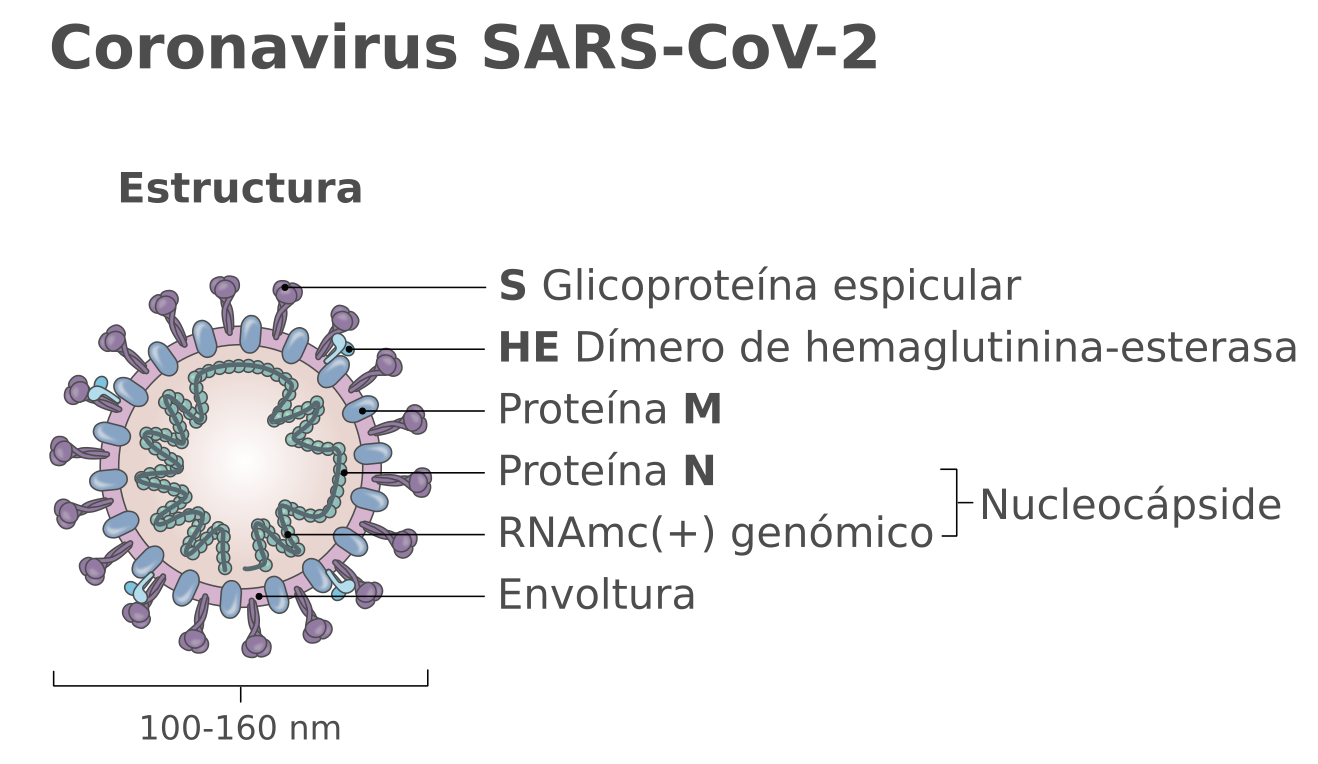
Cuando el virus infecta una célula humana utiliza la maquinaria celular del citoplasma para fabricar las proteínas necesarias para generar nuevos virus, realizar copias de su ARN y ensamblar todas estas piezas en los nuevos viriones que escaparán de la célula para seguir infectando. Durante todo este proceso, ningún componente del virus accede al núcleo celular, que es precisamente donde se encuentra el genoma en forma de ADN, así que este virus lo tendría difícil para integrarse en nuestro genoma, al menos a priori.
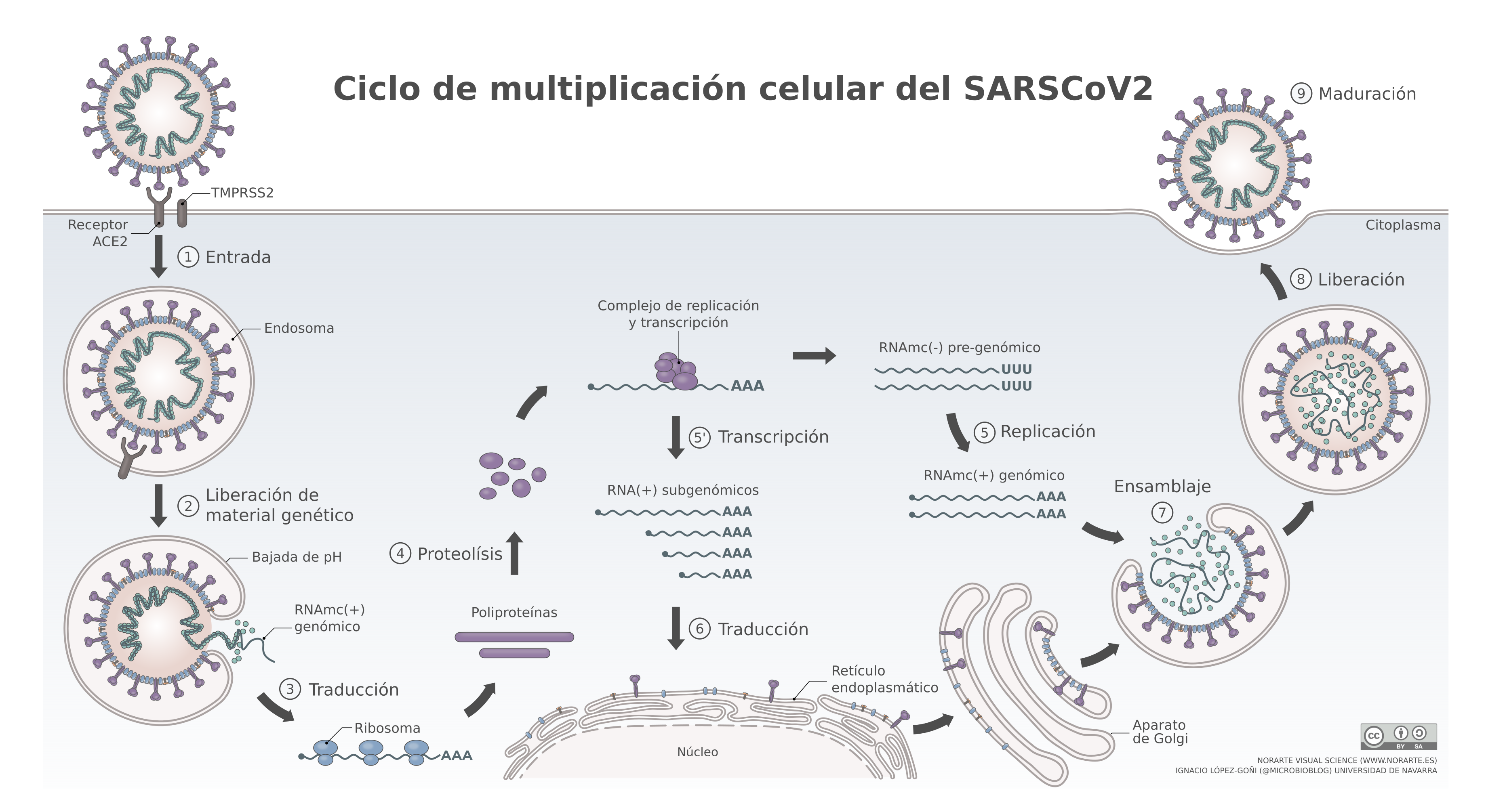
Pero a esta dificultad inicial hay que añadir otra: si algún fragmento del virus se las ingeniara para llegar al núcleo, como su material genético es ARN y el nuestro ADN, tampoco podría integrarse en nuestro genoma sin transformarse previamente a ADN. Excepto si el virus fuera capaz de fabricar una enzima retrotranscriptasa que convirtiera el ARN en ADN (como hacen el VIH y otros retrovirus, que sí se integran en nuestro genoma); pero, por suerte para nosotros, no tiene ningún gen que codifique este tipo de enzimas. Y, por supuesto, el genoma humano no tiene genes para realizar transcripción inversa. ¿O sí?
Ya os he hablado en otras ocasiones de los elementos móviles del ADN humano (también conocidos como retrotransposones) y de su papel en el desarrollo tumoral. Estos elementos serían secuencias de ADN capaces de «saltar» de una posición a otra del genoma, proceso durante el cual se crea una nueva copia de la secuencia en otra posición del genoma a partir de un intermedio de ARN mensajero (ARNm).
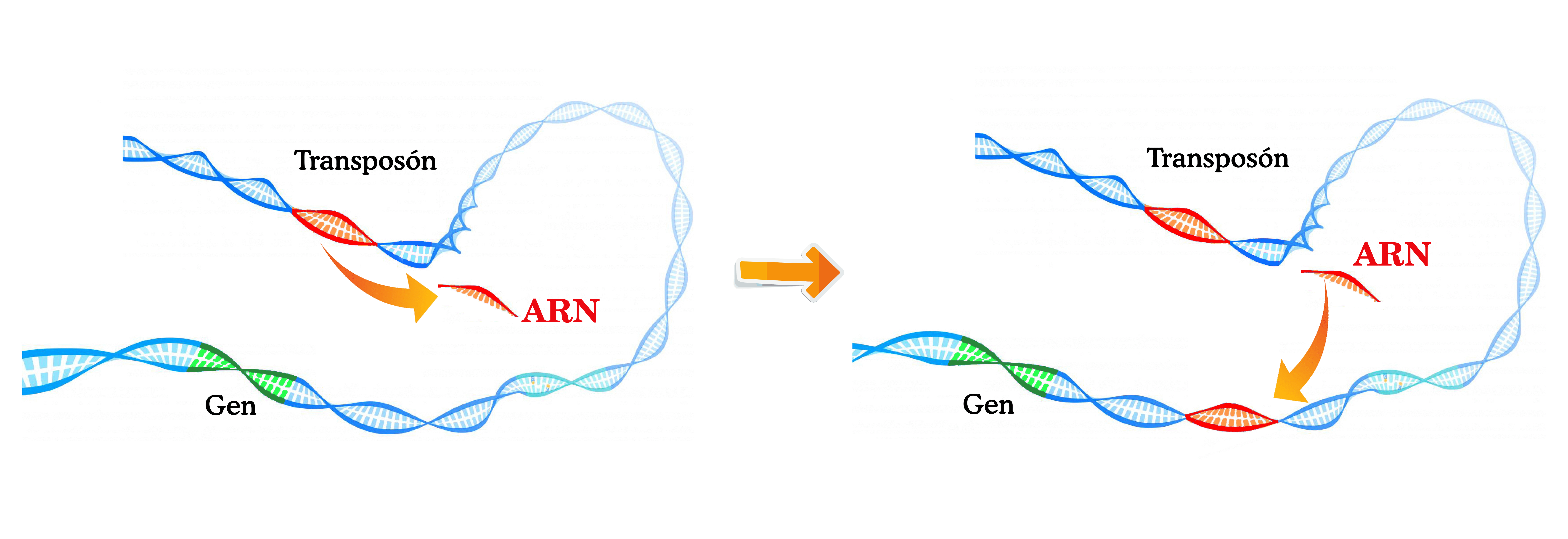
Para dar este salto, estos elementos móviles necesitan dos cosas: por un lado, una retrotranscriptasa que transcriba su ARNm a una copia de ADN; por otro, una enzima que corte el ADN en el lugar de integración para insertarlo allí. Pero justamente uno de estos elementos móviles, el conocido como LINE-1, tiene en su secuencia un gen que realiza ambas dos funciones. Y en nuestro genoma tenemos unas 500 000 copias de LINE-1, de las cuales en torno a un centenar son activas. En resumen, nuestras células sí son capaces de fabricar la enzima retrotranscriptasa que necesita el SARS-CoV-2 para integrar su ARN en nuestro genoma.
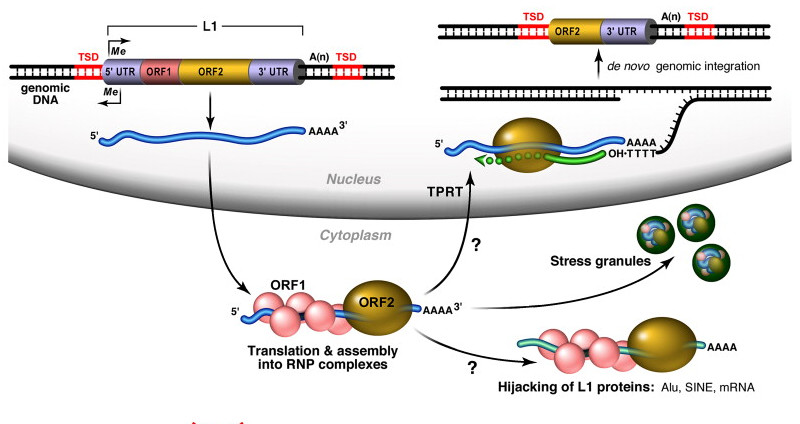
Pero este virus no lo va a tener fácil: en la inmensa mayoría de células de nuestro cuerpo los elementos LINE-1 están reprimidos y apenas pueden expresarse, por lo que su capacidad de fabricar enzima retrotranscriptasa es enormemente baja. Sí hay otros tipos celulares en losl que hay una mayor expresión (células tumorales, embrionarias o neuronales) pero no son relevantes en la infección de COVID-19. Además, incluso en caso de expresión de LINE-1 y fabricación de retrotranscriptasa, este elemento móvil tiene «preferencia» por integrarse a él mismo en el genoma o a otros elementos móviles (como ALU o SVA).
Así que, a priori, la posibilidad de que el SARS-CoV-2 se integre en el genoma de una célula humana infectada usando la retrotranscriptasa de LINE-1 es tremendamente baja. Por eso sorprendió el estudio de Rudolf Jaenisch en el que decía tener evidencias de integración del virus en el genoma celular humano en pacientes de COVID-19.
Análisis de células humanas in vitro
En el estudio se empieza analizando la infección por SARS-CoV-2 de células humanas in vitro (en células HEK293T, derivadas de riñón de embrión humano). Previendo que la cantidad de retrotranscriptasa procedente de LINE-1 sería pequeña, los investigadores decidieron aumentar artificialmente la cantidad de LINE-1 antes de la infección con SARS-CoV-2. Tras la infección secuenciaron y analizaron el genoma encontrando que, efectivamente, algunos fragmentos del virus se habían integrado en el ADN humano. En concreto, detectaron el gen que codifica la proteína N, pero no el de la espícula, que es el usado en las vacunas de ARNm; posiblemente porque el gen de la proteína N se encuentra en un extremo del genoma del virus. Tras encontrar las secuencias de virus en la secuenciación del genoma se comprobó experimentalmente mediante PCR que efectivamente estas secuencias se encontraban en el ADN humano. Esta validación experimental es imprescindible como evidencia.

Así pues, en este experimento sí encontraron fragmentos del virus integrados en el genoma humano, pero fue con células in vitro en las que se había aumentado artificialmente la cantidad de LINE-1. Estos niveles de expresión de LINE-1 son mucho mayores que los que se encuentran en una célula normal de nuestro organismo (salvo casos concretos de células tumorales o embrionarias). Esta fue una de las críticas al artículo publicado en diciembre. Por ello, en la nueva versión del artículo publicada en PNAS analizaron si había inserciones del virus en el genoma de células HEK293T en las que no se había aumentado previamente la cantidad de LINE-1 y, pese a afirmar haberlas encontrado, no informan de que las hayan validado experimentalmente. En resumen, no presentan evidencias suficientes de que el SARS-CoV-2 se integre en el genoma humano sin un aumento previo y artificial de LINE-1. Y esto, obviamente, todavía en células in vitro.
Análisis de células humanas de pacientes infectados
Con tal de comprobar si eran capaces de encontrar si el virus se había integrado en el genoma celular de pacientes con COVID-19, el equipo de investigadores usó bases de datos publicadas con secuenciaciones de ARN de personas infectadas. Es importante destacar que no tuvieron acceso a células reales sino sólo a las secuenciaciones en bases de datos, por lo que no pudieron validar sus hallazgos experimentalmente.
En estas secuenciaciones de ARN buscaron quimeras virus-humano, es decir, fragmentos de ARN en el que una parte de la secuencia fuera humana y otra del SARS-CoV-2. ¿Por qué? Si el virus se hubiera integrado en el genoma humano, al transcribirse el ADN a ARN mensajero, se hallarían fragmentos de ARN con secuencias de ambas especies.

El porcentaje de fragmentos del virus que encontraron se hallaba por debajo del 0.14 %. Según los autores, esto es coherente con una integración del virus en el ADN humano y su transcripción posterior a ARN mensajero (que es lo que detecta la secuenciación de ARN). Y aquí está una de las grandes pegas del estudio, así que vamos a intentar explicarla con calma.
El proceso de secuenciación de ADN ha avanzado enormemente desde la publicación del primer borrador del Proyecto Genoma Humano, hace ya 20 años. Actualmente podemos secuenciar el genoma de una persona (es decir, leer las más de 3 000 millones de letras de su ADN), procesarlo y analizarlo en menos de 24 horas. Pero esta técnica se ha optimizado para la lectura del ADN, no de ARN, así que cuando analizamos el conjunto de secuencias de ARN celular (el denominado transcriptoma) debemos realizar primero la conversión de ARN a ADN (denominado ADN complementario, ADNc). Ya hemos hablado de esto unos párrafos atrás, cuando comentábamos que para poder integrarse el SARS-CoV-2 en nuestro genoma necesitaba primero convertir su material genético a ADN con enzimas retrotranscriptasas. Por ello, para la secuenciación de ARN un primer paso es añadir estas enzimas a la muestra a analizar.
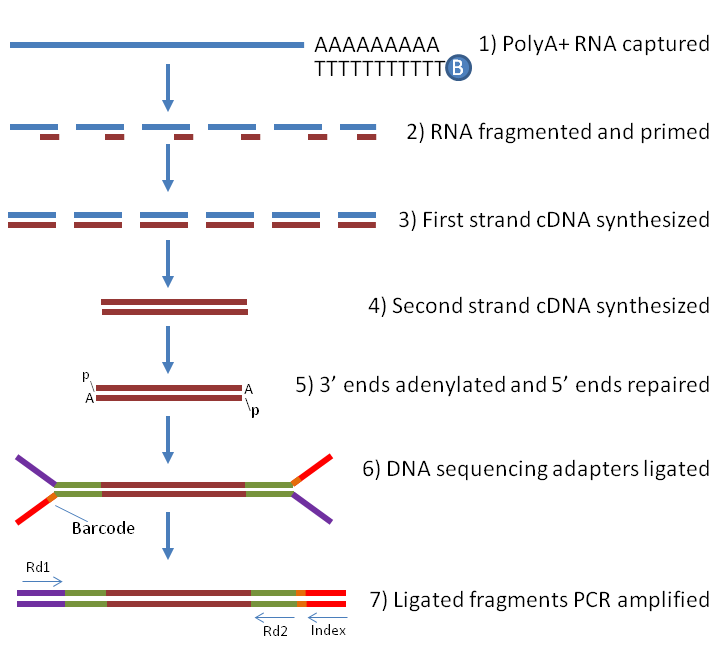
Tal y como se muestra en la imagen anterior, de la secuenciación se obtiene la cadena de nucleótidos de cada uno de las secuencias obtenidas al fragmentar el ARN. Hay que tener en cuenta que en este proceso se analiza todo el ARN celular, así que si hay otro tipo distinto del humano (por ejemplo, procedente de una infección vírica como SARS-CoV-2) también lo encontraremos en nuestro análisis pero, al provenir de ARN inicial distinto (el punto de partida señalado en la figura anterior como (1)) deberíamos localizarlo en fragmentos diferentes: es decir, deberíamos encontrar fragmentos de ARN relacionado con genes humanos o virus, pero no esperar encontrar fragmentos de ARN con mezclas de ambos.
El problema radica que en la técnica de secuenciación de ARN, al transformar mediante enzimas todos los fragmentos de ARN a ADNc, puede ocurrir que se «peguen» fragmentos de ARN de distintas procedencias, así que las quimeras encontradas en el estudio de Jaenisch podrían no ser más que un artefacto de la preparación de las muestras. De hecho, un estudio recientemente publicado en BiorXiv por Yan et al. (y que todavía no se ha publicado en una publicación revisada por pares) estima que en la secuenciación del ARN de una muestra de células humanas de pacientes con COVID-19 se obtienen durante el procesado de las muestras hasta un 1 % de quimeras virus-humano derivadas del propio procedimiento experimental. Es decir, las quimeras que encuentran los autores (recordemos, un 0.14 %) puede que no sean más que errores de la preparación de muestras.
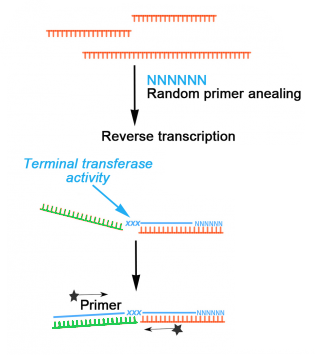
Tras recibir estas críticas en la versión inicial del artículo, los investigadores trataron de buscar en los datos alguna pista que reforzara su teoría de que las quimeras observadas no eran un artefacto del procedimiento experimental y provenían de una integración real del virus SARS-CoV-2 en el genoma humano. Y formularon la siguiente hipótesis: el ARN quimérico procedente de una integración en el ADN celular (que recordemos tiene dos hebras con la misma información pero en sentidos opuestos) daría lugar a quimeras también en ambos sentidos a partes iguales; mientras que si el ARN quimérico procedía de un artefacto experimental (obtenido del virus con una sola hebra en un sentido específico) las quimeras tendrían el ARN del virus en un solo sentido. Un análisis de las muestras de pacientes reveló que, como ellos esperaban, las secuencias de las quimeras tenían el ARN vírico como una mezcla de ambos sentidos.
El problema es que esta idea no es más que una hipótesis. Esta mezcla en distintos sentidos podría deberse a causas todavía no exploradas, por lo que no representa una evidencia suficiente de integración del virus en el genoma celular. Además, hay que recordar que este análisis de muestras de pacientes reales se hizo a partir de secuenciaciones obtenidas de bases de datos online, por lo que no se pudo validar experimentalmente ninguna de las inserciones detectadas.
En resumen, en el estudio de Jaenisch no se demuestra inequívocamente la presencia de integraciones del virus SARS-CoV-2 en el genoma humano, ya que los únicos casos de inserciones validadas experimentalmente se dan en células humanas in vitro en las que se ha sobreexpresado LINE-1 para aumentar la cantidad de enzima retrotranscriptasa. Es decir, reitero, no se ha encontrado experimentalmente que el virus se encuentre integrado en ningún genoma celular humano. Al menos por ahora. Porque se están mejorando las técnicas de secuenciación de ARN para evitar la aparición de quimeras; en el artículo de Yan et al. anteriormente citado ya se propone un método experimental que evita el enriquecimiento de quimeras durante la secuenciación de ARN.
Conclusión
¿Significa esto que es absolutamente imposible que el SARS-CoV-2 se integre en una célula infectada? No, ni mucho menos. El elemento LINE-1, muy de vez en cuando, integra en el genoma celular secuencias de ARNm de cualquier tipo que encuentre en el citoplasma. Y también muy muy muy de vez en cuando lo hace en células germinales, las que dan lugar a espermatozoides y óvulos, las que transmiten esa inserción a su descendencia. Justamente por eso, tenemos nuestro genoma plagado de pseudogenes, copias de fragmentos de genes en una posición distinta a su localización original. Y si pueden integrar muy muy muy de vez en cuando cualquier ARN mensajero, ¿por qué iba a ser distinto con el ARN del SARS-CoV-2? Este artículo polémico demuestra que, en las líneas celulares y sobreexpresando LINE-1, el mecanismo de integración es posible. Estoy convencido de que que se acabarán confirmando integraciones de SARS-CoV-2 tarde o temprano. Pero esa no es la pregunta que nos debemos hacer. Lo que nos interesa es si es relevante, si va afectar a muchas personas (con lo que, como apuntan los autores, podría dar positivos en PCR de personas sanas) o va a ser completamente irrelevante. En mi opinión, no creo que este fenómeno sea excesivamente relevante, pero mientras no se investigue no lo sabremos.
En cuanto al uso que hacen los negacionistas de este artículo, no hagamos caso y no les demos más voz. Así que cuando podáis, vacunaos.
Referencias
- Zhang L, Richards A, Khalil A, Wogram E, Ma H, Young RA, Jaenisch R. SARS-CoV-2 RNA reverse-transcribed and integrated into the human genome. bioRxiv [Preprint], 2020. doi: 10.1101/2020.12.12.422516.
- Zhang L, Richards A, Barrasa M, Hughes SH, Young RA, Jaenisch R. Reverse-transcribed SARS-CoV-2 RNA can integrate into the genome of cultured human cells and can be expressed in patient-derived tissues. Proceedings of the National Academy of Sciences, 118(21), 2021. doi: 10.1073/pnas.2105968118
- Yan B, Chakravorty S, Mirabelli C, Wang L, Trujillo-Ochoa JL, Chauss D, Kumar D, Lionaki MS, Olson MR, Wobus CE, Afzali B, Kazemian M. Host-virus chimeric events in SARS-CoV2 infected cells are infrequent and artifactual. bioRxiv 2021.02.17.431704; doi: 10.1101/2021.02.17.431704
- Cohen J. Further evidence supports controversial claim that SARS-CoV-2 genes can integrate with human DNA. Science, May, 6, 2021. doi: 10.1126/science.abj3287
Sobre el autor: Guillermo Peris es doctor en química cuántica, profesor del Departamento de Lenguajes y Sistemas Informáticos de la Universitat Jaume I e investigador de GENYO (Centro Pfizer – Universidad de Granada – Junta de Andalucía de Genómica e Investigación Oncológica).
Óscar
Hola,
Muchas gracias por el artículo. No tengo ningún tipo de formación en biología, pero de vez en cuando me gusta leer sobre estos temas y tratar de entender el por qué de las cosas. Hay dos temas que no consigo comprender.
– Cuando algunas personas (básicamente antivacunas o conspiranoicos) decían que las vacunas de ARN mensajero podían integrarse en el genoma humano, uno de los argumentos que se empleaban para refutar esta idea era que para poder emplear transcriptasas inversas se necesita un cebador. Por lo que he podido leer, parece que retrovirus como el virus del sida incorporan cebadores dentro del virión. En el caso de los retrotransposones creo recordar que empleaban secuencias del propio ADN del núcleo como cebador. Pero…¿Y para el ARN del sars-cov-2?¿De dónde sacamos el cebador? ¿Puede LINE-1 retrotrascribir ARN del sars-cov-2 sin necesidad de ningún cebador específico del sars-cov-2?
– En diciembre un usuario de twitter publicó un hilo (https://twitter.com/_mariusW/status/1338275889268760576), en el que comentaba lo siguiente: «They say they can find chimeric RNA-seq reads between SARS-2 and the human genome. I would bet a lot of money that these read originate from RNA recombination between SARS-2 and human mRNAs. There is an alternative hypothesis that is in my opinion way more likely. I would bet a lot of money that these read originate from RNA recombination between SARS-2 and human mRNAs. SARS-Cov-2 replication naturally use a complex mechanism of discontinuous transcription that involve a template switch between two different regions of the SARS-2 genome. This is not hard to imagine that errors in template switching would cause the replication complex to jump from SARS-2 to human mRNA, creating chimeric RNA sequences. The good thing is that hypothesis is very easy to test: I predict that all chimeric sequences will originate from exons (or other transcribed regions), and that they will be very few (or none) chimeric reads from introns or intragenic sequences. If these chimeric reads originated from integrated SARS-2 sequences, then you would expect them to originate mostly from intra- or inter-genic regions, and very rarely from exons. The opposite being true if these reads comes from template switching errors» . Si tenemos en cuenta que el gen viral encontrado en adn de la célula HEK293T es el de la proteína N, que está en el extremo del genoma del virus. ¿Este hecho estaría respaldando la teoría del usuario de twitter que referencio?
Muchas gracias.
Hugo
Muy buena información.